hisat2:要想知道这是啥,自行搜索了解。反正是用来建索引和mapping的而且是大家普遍都用的一个生信工具。首先下载二进制编译后的文件。
下载以及解压,如下:
wget ftp://ftp.ccb.jhu.edu/pub/infphilo/hisat2/downloads/hisat2-2.1.0-Linux_x86_64.zipunzip hisat2-2.1.0-Linux_x86_64.zip
然后这个hisat2需要把他的整个hisat2文件夹加入环境变量中。具体操作自己百度。因为我加了也不能用……因此,每次使用hisat2文件夹所在路径指定。
比对前需要建立索引:
hisat2-build -p 4 genome.fa genome#这个建索引一定要进入到你下载好的参考基因组的文件夹里去操作,否则建好了mapping是找不到文件的。#genome是基因组的名字 比如人类是hg19 那么示例代码就是:hisat2-build -p 4 hg19.fa hg19
具体建立索引的时间看物种和电脑配置
建好后会有8个文件。
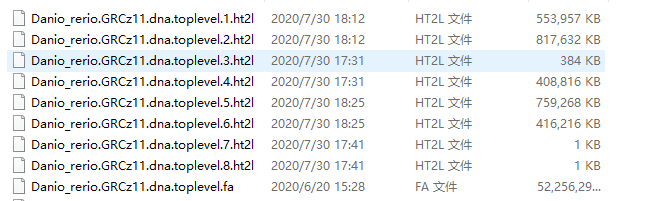
开始mapping
hisat2 -x hg19 -p 20 -U reads.fq -S align.sam# -U单端测序数据输入 -x 后跟索引所在的文件路径/位置。-p 后接线程数 -U 后接输入的测序序列位置-S 表示输出格式指定为sam文件 align.sam 输出序列名+后缀名sam
hisat2 -x hg19 -p 20 -1 R1.fq -2 R2.fq -S align.sam#双端测序的使用方法 分别用-1 -2 指定两端的输入
举一个个人的例子:
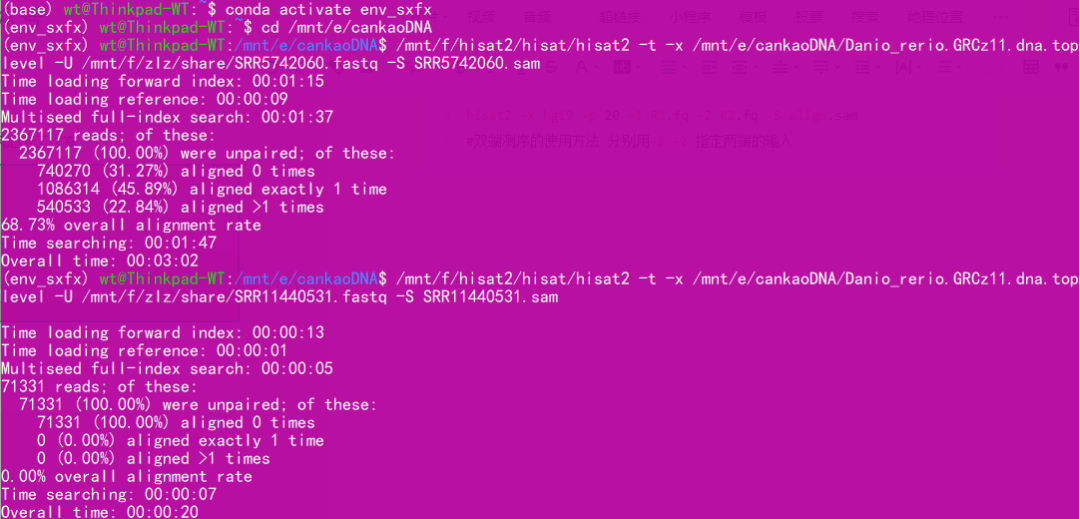
完成后会生成sam文件
具体更多的用法可在电脑上进入hisat2文件夹后,用./hisat2 测试是否可用以及查看用法,获取更多参数信息请登录官网查看。
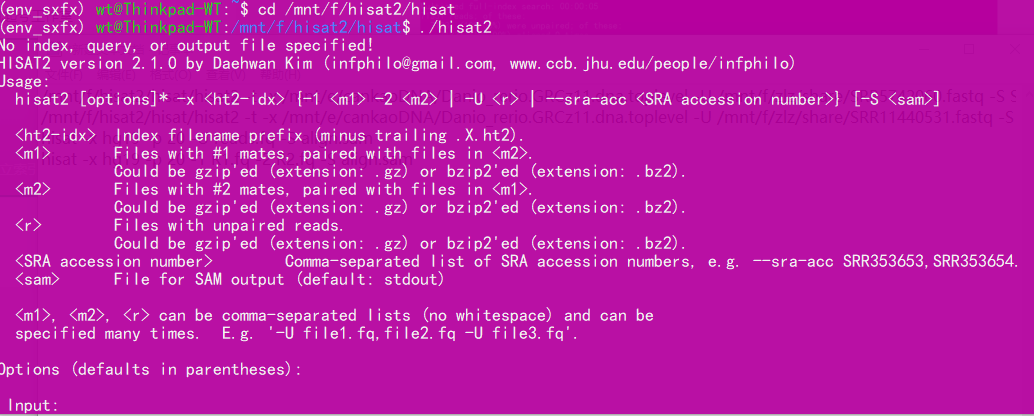
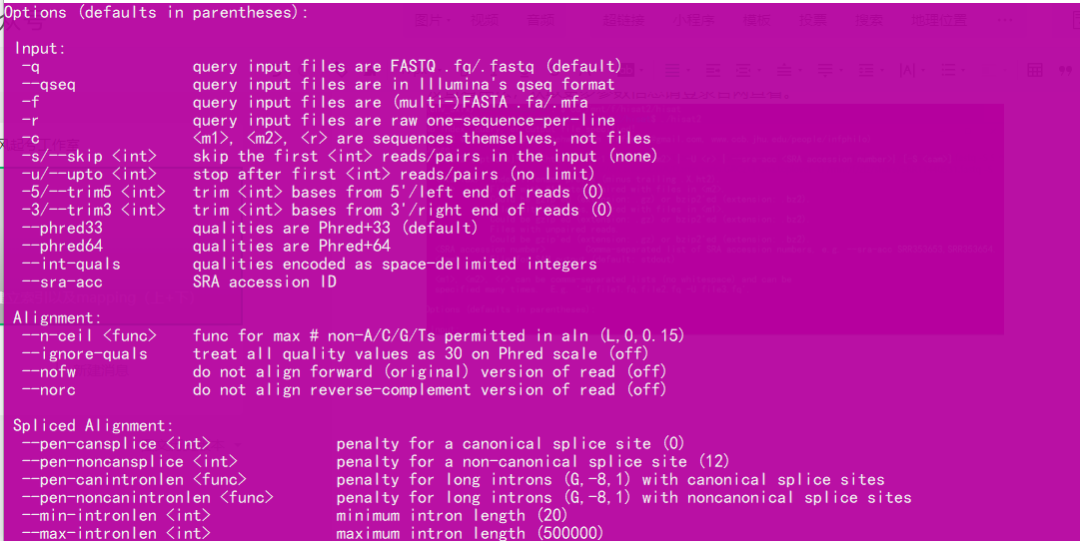
本文内容由网友自发贡献,版权归原作者所有,本站不承担相应法律责任。如您发现有涉嫌抄袭侵权的内容,请联系:hwhale#tublm.com(使用前将#替换为@)