Alpha 多样性指数
常见的Alpha多样性指数的含义见https://blog.csdn.net/rojyang/article/details/81326378
R代码如下:
### library(vegan) ##加载群落数据分析常用的包
data(BCI) ##vegan包中自带的群落数据
shannon <- diversity(BCI,MARGIN = 1) ## Shannon 多样性指数, MARGIN=1 计算行的多样性 ,一般的群落数据表行名为样方名,列名为物种名 index默认为“shannon”
simpson <- diversity(BCI,index = "simpson",MARGIN = 1) ## Simpson多样性指数
SR <- specnumber(BCI,MARGIN = 1) ## 物种丰富度:相当于每一行大于0 (生物量、盖度或多度) 的物种数
Pielou <- shannon/log(SR) ##均匀度指数
diversity <- data.frame(shannon,simpson,SR,Pielou) ##将物种丰富度、Shannon多样性指数、Simpson多样性指数和Pielou' 均匀度指数放置在一个数据框中
write.csv(diversity,"diversity.csv") ## 将多样性结果导出为csv文件
Beta多样性指数
Hill的Beta多样性指数和Gamma多样性指数
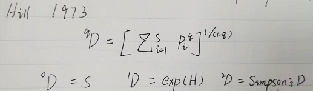
library(vegan)
library(vegetarian)
data(dune)
d(dune,lev = "alpha",q=0) ## 平均alpha 物种丰富度
d(dune,lev = "alpha",q=1) ## 平均alpha Shannon多样性
d(dune,lev = "alpha",q=2) ## 平均alpha Simpson多样性
d(dune,lev = "beta",q=0) ##倍性beta物种丰富度
d(dune,lev = "beta",q=1) ##倍性beta Shannon多样性
d(dune,lev = "beta",q=2) ##倍性beta Simpson多样性
d(dune,lev = "gamma",q=0) ##Gamma物种丰富度
d(dune,lev = "gamma",q=1) ##Gamma Shannon多样性
d(dune,lev = "gamma",q=2) ##Gamma Simpson多样性
基于距离和方差分解的Beta多样性指数
library(vegan)
betadiver(dune,method = "w") ## The basic Whittaker index
betadiver(help=TRUE) ## 共有24中计算Beta多样性(距离)的方法
library(adespatial)
dune.beta <- beta.div(dune,method = "hellinger",nperm = 999)
dune.beta$beta
dune.beta$SCBD ## 大于平均值的物种
dune.beta$LCBD ##Beta 多样性样方贡献
功能多样性
单性状(混合性状)功能多样性
data(dummy)
ex1 <- dbFD(dummy$trait, dummy$abun) ##混合性状或单一性状的功能多样性
ex1
多性状多样方功能多样性
library(tidyverse)
library(FD)
## 制作空数据框用于后续存放数据
Plot<- 'abc'
trait1<-0
trait2<-0
trait3<-0
trait4<-0
trait5<-0
trait6<-0
trait7 <- 0
trait8 <- 0
CWM.cover.result <- data.frame(Plot,trait1,trait2,trait3,trait4,trait5,trait6,trait7,trait8)
CWM.cover.result$Plot <- as.character(CWM.cover.result$Plot)
str(CWM.cover.result)
FDis.cover.result <- CWM.cover.result
Trait <- c('trait1','trait2','trait3','trait4','trait5','trait6','trait7','trait8')
Plot <- unique(trait_mean$Plot)
#用循环来计算每个样方每种性状的功能多样性
#cycle
for (i in 1:8) {
for (v in 1:72) {
Trait.i <- Trait[i] ## 第i个性状
Plot.v <- Plot[v] ## 第v个样方
Data.i <- trait_mean%>% ## 选择性状数据
dplyr::select('Plot','Species',Trait.i)%>%
filter(Plot==Plot.v)
names(Data.i)<-c('Plot','Species','Trait_i')
Data.i<-Data.i%>%
filter(Trait_i != 'NaN')
Cover<-cover%>% ## 选择群落数据(这里使用盖度数据) 长数据
filter(Species %in% unique(Data.i$Species))%>%
filter(Plot==Plot.v)
Data.i<- Data.i%>%
filter(Species %in% unique(Data.i$Species))
data.db <-merge(Data.i,Cover,by=c('Plot','Species'))%>% ## 将两种数据组合
filter(Cover != 0)
data.db.trait <- dplyr::select(data.db,Species,Trait_i)
row.names(data.db.trait) <- data.db.trait$Species
data.db.trait<-subset(data.db.trait,select = Trait_i)
data.db.cover <- dplyr::select(data.db,Species,Cover)%>% ## 将盖度长数据转化为宽数据
spread(key=Species,value=Cover)
CWM <- dbFD(x=data.db.trait, a=data.db.cover, calc.FRic = F, calc.FDiv = F, w.abun = T, calc.CWM = TRUE) ## 计算功能多样性,这里主要计算了FDis和CWM
CWM.cover.result[v,1] <- Plot.v
CWM.cover.result[v,i+1] <- CWM$CWM[1,1]
FDis.cover.result[v,1] <- Plot.v
FDis.cover.result[v,i+1] <- CWM$FDis
}
}
CWM.cover.result <- CWM.cover.result%>% ## 将结果转化为长数据格式并重命名
gather(key=key,value = value,-Plot)%>%
mutate(CWM='CWM')%>%
unite(key2,CWM,key,sep="_")%>%
spread(key=key2,value = value)
FDis.cover.result <- FDis.cover.result%>% ## 将结果转化为长数据格式并重命名
gather(key=key,value = value,-Plot)%>%
mutate(CWM='FDis')%>%
unite(key2,CWM,key,sep="_")%>%
spread(key=key2,value = value)
系统发育多样性
com_2020 <- read.csv("com2020.csv",row.names = 1) ## 读取群落数据
library(ape)
library(picante) ## 加载系统发育分析包
phylo <- read.tree("tree.treefile") ## 读取系统发育树文件
rooted <- root(phylo,outgroup = "Amborella_trichopoda",resolve.root = T) ## 转化为有根树
a <- cophenetic(rooted)%>%as.data.frame() ## 转化为距离矩阵
cover2020.pd<-pd(com_2020,rooted) ## 求Fish' 系统发育距离
cover2020.mpd.ab<-mpd(com_2020,a,abundance.weighted = FALSE) ## 平均系统发育距离
cover2020.mntd.ab<-mntd(com_2020,a,abundance.weighted = FALSE) ## 平均最近种系统发育距离
cover2020.mpd.ab<-mpd(com_2020,a,abundance.weighted = TRUE) ## 平均系统发育距离 (盖度加权)
cover2020.mntd.ab<-mntd(com_2020,a,abundance.weighted = TRUE) ## 平均最近种系统发育距离 (盖度加权)
cover2020.ses.mpd.pa<-ses.mpd(com_2020,a,abundance.weighted = FALSE) ## 标准化平均系统发育距离 物种池为系统发育树物种池
cover2020.ses.mntd.pa<-ses.mntd(com_2020,a,abundance.weighted = FALSE) ## 标准化平均最近种系统发育距离
cover2020.ses.mpd.pa<-ses.mpd(com_2020,a,abundance.weighted = FALSE) ## 标准化平均系统发育距离 (盖度加权)
cover2020.ses.mntd.pa<-ses.mntd(com_2020,a,abundance.weighted = FALSE) ## 标准化平均最近种系统发育距离 (盖度加权)
参考文献:
Fisher, R.A., Corbet, A.S. & Williams, C.B. (1943). The relation between the number of species and the number of individuals in a random sample of animal population. Journal of Animal Ecology 12, 42–58.
Hurlbert, S.H. (1971). The nonconcept of species diversity: a critique and alternative parameters. Ecology 52, 577–586.
Jost, L. 2006. Entropy and diversity. Oikos 113(2): 363-375.
Jost, L. 2007. Partitioning diversity into independent alpha and beta components. Ecology 88(10): 2427-2439.
Hill, M. 1973. Diversity and evenness: A unifying notation and its consequences. Ecology 54: 427-432.
Baselga, A. (2010) Partitioning the turnover and nestedness components of beta diversity. Global Ecology and Biogeography 19, 134–143.
Koleff, P., Gaston, K.J. and Lennon, J.J. (2003) Measuring beta diversity for presence-absence data. Journal of Animal Ecology 72, 367–382.
Marion, Z.H., Fordyce, J.A. and Fitzpatrick, B.M. (2017) Pairwise beta diversity resolves an underappreciated source of confusion in calculating species turnover. Ecology 98, 933–939.
Whittaker, R.H. (1960) Vegetation of Siskiyou mountains, Oregon and California. Ecological Monographs 30, 279–338.
Chao, A., R. L. Chazdon, R. K. Colwell and T. J. Shen. 2006. Abundance-based similarity indices and their estimation when there are unseen species in samples. Biometrics 62: 361-371.
Legendre, P. 2014. Interpreting the replacement and richness difference components of beta diversity. Global Ecology and Biogeography 23: 1324-1334.
Legendre, P. and D. Borcard. 2018. Box-Cox-chord transformations for community composition data prior to beta diversity analysis. Ecography 41: 1820-1824.
Legendre, P. and M. De Cáceres. 2013. Beta diversity as the variance of community data: dissimilarity coefficients and partitioning. Ecology Letters 16: 951-963.
Legendre, P. and E. D. Gallagher, E.D. 2001. Ecologically meaningful transformations for ordination of species data. Oecologia 129: 271-280.
Legendre, P. and Legendre, L. 2012. Numerical Ecology. 3rd English edition. Elsevier Science BV, Amsterdam.
Webb, C., D. Ackerly, M. McPeek, and M. Donoghue. 2002. Phylogenies and community ecology. Annual Review of Ecology and Systematics 33:475-505.