需要安装的工具包
pip install numpy pandas matplotlib seaborn wheel pandas_profiling jupyter notebook -i https://pypi.tuna.tsinghua.edu.cn/simple
pip install graphviz pydotplus -i https://pypi.tuna.tsinghua.edu.cn/simple
后面需要画出决策树,需要将将graphviz加入系统环境变量当中,以及将如下代码加入程序中。
import os
os.environ["PATH"]+=os.pathsep+"G:/soft_exe/Graphviz/bin/"
"G:/soft_exe/Graphviz/bin/"是你的graphviz的安装目录。
安装机器学习第三方工具包
pip install scikit-learn -i https://pypi.tuna.tsinghua.edu.cn/simple
安装机器学习可解释性第三方工具包
pip install pdpbox eli5 -i https://pypi.tuna.tsinghua.edu.cn/simple
安装机器学习可解释性分析工具shap
pip install shap -i https://pypi.tuna.tsinghua.edu.cn/simple
在安装shap时候,可能会出现缺少MICROSOFT VISUAL C++ 14.0的问题:我尝试了几种方案,然后最后解决的是通过安装vs解决的,如下图那个选项一定要选。

数据简单探索
import pandas as pd
df=pd.read_csv("heart.csv")
df.head()
数据集的话,建议大家注册一个账号去kaggle上面下载就好了。

查看数据集大小
df.shape

查看各个列的名
df.columns

df.info()

查看各个列是否有缺失值
df.isnull().sum()

一行代码生成探索性分析EDA报告
import pandas_profiling
profile=pandas_profiling.ProfileReport(df)
profile


基本通过这个探索性报告就能详细了解这个数据集了。这个很强大。
将这个报告保存到本地。
profile.to_file("profile.html")
数据可视化
绘制相关系数矩阵、散点图、直方图、KDE概率密度曲线,小提琴图,柱状图
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
import seaborn as sns
df=pd.read_csv("heart.csv")
df.corr()

plt.figure(figsize=(15,15))
sns.heatmap(df.corr(),annot=True,fmt=".1f",square=True)
plt.show()

sns.pairplot(df)
plt.show()

单个特征统计分布分析
sns.distplot(df['age'])
plt.show()

sns.countplot(x="target",data=df,palette='bwr')
plt.show()

pd.crosstab(df.age,df.target).plot(kind='bar',figsize=(20,6))
plt.title("HeartDiseaseAndAge")
plt.xlabel("Age")
plt.ylabel("Frequency")
plt.savefig("HeartDiseaseAndAge.png")
plt.show()

sns.boxplot(x=df["target"],y=df["age"])
plt.show()

sns.violinplot(x=df["target"],y=df["age"])
plt.show()

不同性别,患病与不患病的分布
pd.crosstab(df.sex,df.target).plot(kind="bar",figsize=(15,6),color=['#1CA53B',"#AA1111"])
plt.title("HeartDiseaseAndAge")
plt.xlabel("Sex")
plt.xticks(rotation=0)
plt.legend(["Haven not Disease","Have Disease"])
plt.ylabel("Frequency")
plt.savefig("HeartDiseaseAndAge.png")
plt.show()

不同心电图特征下,患病与不患病的分布
pd.crosstab(df.slope,df.target).plot(kind="bar",figsize=(15,6),color=['#DAF7A6',"#FF5733"])
plt.title("HeartDiseaseAndSlope")
plt.xlabel("The Slope of the peak exercise St segment")
plt.xticks(rotation=0)
plt.legend(["Haven not Disease","Have Disease"])
plt.ylabel("Frequency")
plt.show()

不同空腹血糖水平,患病与不患病的分布
pd.crosstab(df.fbs,df.target).plot(kind="bar",figsize=(15,6),color=['#FFC300',"#581845"])
plt.title("HeartDiseaseAndFBS")
plt.xlabel("FBS-(Fasting Blood Sugar >120 mg/dl) (1=true;0=false)")
plt.xticks(rotation=0)
plt.legend(["Haven not Disease","Have Disease"])
plt.ylabel("Frequency of Disease or Not")
plt.show()

不同心绞痛的类型,患病和不患病的类型
pd.crosstab(df.cp,df.target).plot(kind="bar",figsize=(15,6),color=['#11A5AA',"#AA1190"])
plt.title("HeartDiseaseAndChestPainType")
plt.xlabel("Chest Pain Type")
plt.xticks(rotation=0)
plt.legend(["Haven not Disease","Have Disease"])
plt.ylabel("Frequency of Disease or Not")
plt.show()

散点图
不同年龄段,不同心率,患病和不患病的分布
plt.scatter(x=df.age[df.target==1],y=df.thalach[df.target==1],c="red")
plt.scatter(x=df.age[df.target==0],y=df.thalach[df.target==0],c="blue")
plt.legend(["Haven Disease","Haven not Disease"])
plt.xlabel("Age")
plt.ylabel("Maximum Heart Rate")
plt.show()

sns.violinplot(x="target",y="age",hue="sex",data=df,split=True)
plt.show()

数据预处理
df.dtypes

df.columns=['age','sex','chest_pain_type','resting_blood_pressure','cholesterol','fasting_blood_sugar','rest_ecg','max_heart_rate_achieved',
'exercise_induces_angina','st_depression','st_slope','num_major_vessels','thalassemis','target']
df.head()
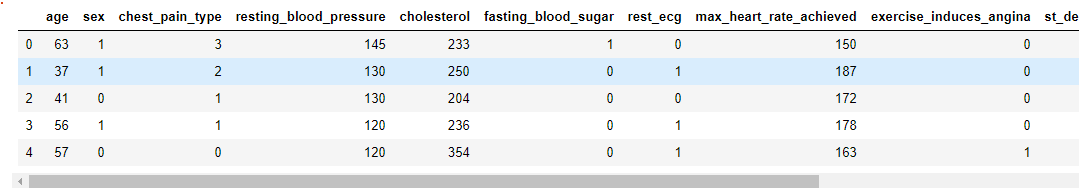
df['sex'][df['sex']==0]='female'
df['sex'][df['sex']==1]='male'
df['chest_pain_type'][df['chest_pain_type']==0]='typical angina'
df['chest_pain_type'][df['chest_pain_type']==1]='atypical angina'
df['chest_pain_type'][df['chest_pain_type']==2]='non-anginal pain'
df['chest_pain_type'][df['chest_pain_type']==3]='asymptomatic'
df['fasting_blood_sugar'][df['fasting_blood_sugar']==0]='lower than 120mg/ml'
df['fasting_blood_sugar'][df['fasting_blood_sugar']==1]='greater than 120mg/ml'
df['rest_ecg'][df['rest_ecg']==0]='normal'
df['rest_ecg'][df['rest_ecg']==1]='ST-T wave abnormality'
df['rest_ecg'][df['rest_ecg']==2]='left ventricular hypertrophy'
df['exercise_induces_angina'][df['exercise_induces_angina']==0]='no'
df['exercise_induces_angina'][df['exercise_induces_angina']==1]='yes'
df['st_slope'][df['st_slope']==0]='upsloping'
df['st_slope'][df['st_slope']==1]='flat'
df['st_slope'][df['st_slope']==2]='downsloping'
df['thalassemis'][df['thalassemis']==0]='unknown'
df['thalassemis'][df['thalassemis']==1]='normal'
df['thalassemis'][df['thalassemis']==2]='fixed defect'
df['thalassemis'][df['thalassemis']==3]='reversable defect'
df.head()

转换之后:
df.dtypes

转化成独热编码格式:
df=pd.get_dummies(df)
df.columns

df.head()

df.iloc[0]

将处理好的数据导出为csv文件。
df.to_csv('process_heart.csv',index=False)
使用pdpbox工具包,对数据集进行探索性数据分析
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
%matplotlib inline
import warnings as wn
wn.filterwarnings("ignore")
df=pd.read_csv('process_heart.csv')
from pdpbox import pdp,get_dataset,info_plots
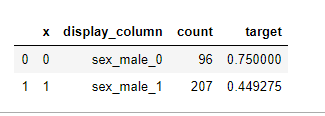
绘制不同性别患病的比例(先验概率统计)
fig,axes,summary_df=info_plots.target_plot(df=df,feature='sex_male',feature_name='gender',target=['target'])
_ = axes['bar_ax'].set_xticklabels(['Female','Male'])

summary_df
心脏周围大血管个数列:患病和不患病的比例
fig,axes,summary_df=info_plots.target_plot(df=df,feature='num_major_vessels',feature_name='num_vessels',target=['target'])

地中海贫血症,患病与不患病的比率
fig,axes,summary_df=info_plots.target_plot(df=df,feature='thalassemis_reversable defect',feature_name='thalassemia_reversable defect',target=['target'])

特征是年龄,患病与不患病的比率
fig,axes,summary_df=info_plots.target_plot(df=df,feature='age',feature_name='age',target=['target'])

不同的最大心率,患病的比例
fig,axes,summary_df=info_plots.target_plot(df=df,feature='max_heart_rate_achieved',feature_name='max_heart_rate_achieved',target=['target'])

分析特征两两交互的影响
feat_name1='num_major_vessels'
nick_name1='num_vessels'
feat_name2='max_heart_rate_achieved'
nick_name2='max_hart_rate'
fig,axes,summary_df=info_plots.target_plot_interact(df=df,features=[feat_name1,feat_name2],feature_names=[nick_name1,nick_name2],target='target')
axes['value_ax'].set_xticklabels(['0','1','2'])
plt.show()

feat_name1='age'
feat_name2='max_heart_rate_achieved'
fig,axes,summary_df=info_plots.target_plot_interact(df=df,features=[feat_name1,feat_name2],feature_names=[feat_name1,feat_name2],target='target')
plt.show()

划分特征列和标签列
数据集
X=df.drop('target',axis=1)
X.shape
标签:
y=df['target']
y.shape
划分训练集和测试集
from sklearn.model_selection import train_test_split
X_train,X_test,y_train,y_test=train_test_split(X,y,test_size=0.2,random_state=10)
X_train.head()

构建随机森林分类模型,在训练集上训练模型
from sklearn.ensemble import RandomForestClassifier
model=RandomForestClassifier(max_depth=5,n_estimators=100,random_state=5)
model.fit(X_train,y_train)

可视化随机森林中的一个决策树
len(model.estimators_)
estimator=model.estimators_[7]
estimator

feature_names=X_train.columns
y_train_str=y_train.astype("str")
y_train_str[y_train_str=='0']='no disease'
y_train_str[y_train_str=='1']='disease'
y_train_str=y_train_str.values
import os
os.environ["PATH"]+=os.pathsep+"G:/soft_exe/Graphviz/bin/"
from sklearn.tree import export_graphviz
export_graphviz(estimator,out_file='tree.dot',feature_names=feature_names,
class_names=y_train_str,
rounded=True,proportion=True,
label='root',precision=2,filled=True
)
from subprocess import call
call(['dot','-Tpng','tree.dot','-o','tree.png','-Gdpi=600'])
from IPython.display import Image
Image(filename='tree.png')

使用eli5工具包,查看各个列的权重
import eli5
eli5.show_weights(estimator,feature_names=feature_names.to_list())


对模型的特征重要性分析
model.feature_importances_

对特征进行排序根据重要性
print("特征排序")
feature_names=X_test.columns
feature_importances=model.feature_importances_
indices=np.argsort(feature_importances)[::-1]
for index in indices:
print("feature %s (%f)"%(feature_names[index],feature_importances[index]))

import eli5
eli5.show_weights(model,feature_names=feature_names.to_list())

对特征权重进行可视化。
plt.figure(figsize=(16,8))
plt.title("feature weight")
plt.bar(range(len(feature_importances)),feature_importances[indices],color="g")
plt.xticks(range(len(feature_importances)),np.array(feature_names)[indices],color="g",rotation=90)
plt.show()

使用模型进行预测
model.predict(X_test)

model.predict_proba(X_test)

查看患病的概率
model.predict_proba(X_test)[:,1]

y_pred=model.predict(X_test)
y_pred_proba=model.predict_proba(X_test)
混淆矩阵
得到混淆矩阵
from sklearn.metrics import confusion_matrix
confusion_matrix_model=confusion_matrix(y_test,y_pred)
confusion_matrix_model
混淆矩阵可视化模板
import itertools
def cnf_matrix_plotter(cm,classes):
plt.imshow(cm,interpolation='nearest',cmap=plt.cm.Oranges)
plt.title("Confusion Matrix")
plt.colorbar()
tick_marks=np.arange(len(classes))
plt.xticks(tick_marks,classes,rotation=45)
plt.yticks(tick_marks,classes)
threshold=cm.max()/2
for i,j in itertools.product(range(cm.shape[0]),range(cm.shape[1])):
plt.text(j,i,cm[i,j],horizontalalignment="center",color="white" if cm[i,j] > threshold else "black",fontsize=25)
plt.tight_layout()
plt.ylabel("True Label")
plt.xlabel("Predict Label")
plt.show()
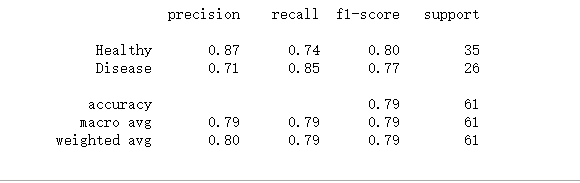
cnf_matrix_plotter(confusion_matrix_model,["Healthy",'Disease'])

from sklearn.metrics import classification_report
print(classification_report(y_test,y_pred,target_names=["Healthy","Disease"]))
Roc曲线
model.predict_proba(X_test)

y_pred_quant=model.predict_proba(X_test)[:,1]
y_pred_quant

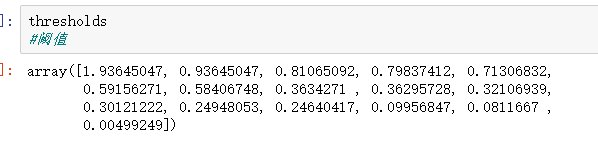
from sklearn.metrics import roc_curve,auc
fpr,tpr,thresholds=roc_curve(y_test,y_pred_quant)

可视化:
plt.plot(fpr,tpr)
plt.plot([0,1],[0,1],ls="--",c=".3")
plt.xlim([0.0,1.0])
plt.ylim([0.0,1.0])
plt.rcParams["font.size"]=12
plt.title("ROC curve")
plt.xlabel("False Positive Rate (1-Specificity)")
plt.ylabel("True Positive Rate(Sensitivity)")
plt.grid(True)

auc(fpr,tpr)
可解释性分析
使用eli5,绘制Permutation Importance图()
Permutation Importance图:对测试集中某一列数据打乱,再将我们打乱的数据集在模型上预测。打乱后是否会导致模型预测精度大幅度下降。
如果大幅度下降,就说这些列度对模型比较重要。
如果没有大幅度下降,甚至更高,这些列可能有噪声,或者列对模型没有作用。
import eli5
from eli5.sklearn import PermutationImportance
perm=PermutationImportance(model,random_state=1).fit(X_test,y_test)
eli5.show_weights(perm,feature_names=X_test.columns.to_list())

上面的绿色的列是对模型来说比较重要的。
Permutation Importance图就是看哪个特征对模型有重要影响。
可解释性分析2
使用pdpbox,绘制PDP图,ICE图,双变量PDP图,可以直观看出每个特征值的不同值对模型预测结果的影响,以及特征交互对模型预测结果的影响。
base_features=df.columns.values.tolist()
base_features.remove("target")
base_features

pdp图(部份依赖图)
pdp图反映了某一个特征在不同值变化时对模型预测结果的影响
注意对比PDP图和先验数据集不同类别分别的差异
from pdpbox import info_plots,get_dataset,pdp,get_dataset
fig,axes,summary_df=info_plots.actual_plot(model=model,X=X_train,feature='sex_male',feature_name='gender',predict_kwds={})

这里指的是女性变成男性,模型预测出患病的概率会降低。
原始数据集中,性别特征统计分布,及患病和不患病的类别分布图。
fig,axes,summary_df=info_plots.target_plot(df=df,feature='sex_male',feature_name='gender',target=['target'])
_=axes['bar_ax'].set_xticklabels(["Female","Male"])

对比,原始数据集,女性患病的概率为0.75,男性患病的概率为0.449,但是这是先验的情况。这里可以直接概率求出。最上面那个是将模型考虑进来,这里可以得出结论男性比女性患病概率低,但是上面那个先验的不能作为因果的结论,不能说统计出来的结果就是男性比女性的概率低。
心脏周围大血管个数:
fig,axes,summary_df=info_plots.actual_plot(model=model,X=X_train,feature='num_major_vessels',feature_name='num_major_vessels',predict_kwds={})

上面也是,将模型考虑进来,大血管数越多,患病的概率越低。
fig,axes,summary_df=info_plots.target_plot(df=df,feature='num_major_vessels',feature_name='num_major_vessels',target=['target'])

最大心率:
fig,axes,summary_df=info_plots.actual_plot(model=model,X=X_train,feature='max_heart_rate_achieved',feature_name='max_heart_rate_achieved',predict_kwds={})

心率越高,模型预测患病的概率也就越高
fig,axes,summary_df=info_plots.target_plot(df=df,feature='max_heart_rate_achieved',feature_name='max_heart_rate_achieved',target=['target'])

ICE图
ice图:将测试集每一个样本在某一个特征变化时候的预测结果显示出来。
对性别特征来说:
feat_name='sex_male'
nick_name='sex'
pdp_dist=pdp.pdp_isolate(model=model,dataset=X_test,model_features=base_features,feature=feat_name)
fig,axes=pdp.pdp_plot(pdp_dist,nick_name,plot_lines=True,frac_to_plot=0.8,plot_pts_dist=True)

上面就是特征由女性变成男性,每一个样本预测患病的概率会降低。
大血管个数:
feat_name='num_major_vessels'
nick_name='num_major_vessels'
pdp_dist=pdp.pdp_isolate(model=model,dataset=X_test,model_features=base_features,feature=feat_name)
fig,axes=pdp.pdp_plot(pdp_dist,nick_name,plot_lines=True,frac_to_plot=0.8,plot_pts_dist=True)

就是大血管数目大量变化的时候,对模型的预测结果可能有什么影响.
feat_name='age'
nick_name='age'
pdp_dist=pdp.pdp_isolate(model=model,dataset=X_test,model_features=base_features,feature=feat_name)
fig,axes=pdp.pdp_plot(pdp_dist,nick_name,plot_lines=True,frac_to_plot=0.8,plot_pts_dist=True)

最大心率对预测结果的影响
feat_name='max_heart_rate_achieved'
nick_name='max_heart_rate_achieved'
fig,axes,summary_df=info_plots.target_plot(df=df,feature=feat_name,feature_name=nick_name,show_percentile=True,target=['target'])

fig,axes,summary_df=info_plots.actual_plot(model=model,X=X_train,feature="max_heart_rate_achieved",feature_name="max_heart_rate_achieved",predict_kwds={})

上面是pdp图的分布,pdp图是考虑了模型的。
pdp_dist=pdp.pdp_isolate(model=model,dataset=X_test,model_features=base_features,feature='max_heart_rate_achieved')
fig,axes=pdp.pdp_plot(pdp_dist,'max_heart_rate_achieved')

feat_name="max_heart_rate_achieved"
nick_name="max_heart_rate_achieved"
pdp_dist=pdp.pdp_isolate(model=model,dataset=X_test,model_features=base_features,feature=feat_name)
fig,axes=pdp.pdp_plot(pdp_dist,'max_heart_rate_achieved',plot_lines=True,frac_to_plot=0.8,plot_pts_dist=True)

二维的pdp图(表达特征之间的交互关系)
feat_name1="max_heart_rate_achieved"
nick_name1="max_heart_rate_achieved"
feat_name2="num_major_vessels"
nick_name2="num_major_vessels"
inter1=pdp.pdp_interact(model=model,dataset=X_test,model_features=base_features,features=[feat_name1,feat_name2])
fig,axes=pdp.pdp_interact_plot(pdp_interact_out=inter1,feature_names=[nick_name1,nick_name2],plot_type="contour",x_quantile=True,plot_pdp=True)

心率越高,血管越少。患病概率高。
fig,axes=pdp.pdp_interact_plot(inter1,[nick_name1,nick_name2],plot_type="grid",x_quantile=True,plot_pdp=True)

这个是带有概率的。
计算测试集每个样本的每个特征对两类预测结果的shape值
import shap
shap.initjs()
explainer=shap.TreeExplainer(model)
shap_values=explainer.shap_values(X_test)
shap_values[1].shape
对测试集所有样本,预测为患病和不患病各自的平均概率
explainer.expected_value

对某个样本,模型预测为患病的概率为测试集患病的平均概率和该样本各特征对患病预测结果的shap值之和。
对某个样本,模型预测患病的概率就是explainer.expected_value[1]与该样本各个特征shap值之和。
Shap值可视化分析
特征重要度
对于某个特征,计算测试集每个病人的该特征shap值之和,shap值越高,特征越重要。
shap.summary_plot(shap_values[1],X_test,plot_type='bar')

已经用过三种构建特征重要度的方法了。使用weight,PermutationImportance和shap这三种方式衡量特征重要度.
各个特征的数值大小和各特征的shap值关系图
summary plot为每个样本绘制其每个特征的shap值,每一行代表一个特征,横坐标为shap值。一个点代表一个样本,颜色表示特征值(红色高,蓝色低)
每一行表示一个特征,红色表示该特征的值较高的数据点,蓝色表示该特征较低的数据点
红色是正向贡献,蓝色是负向贡献.
shap.summary_plot(shap_values[1],X_test)

shap.summary_plot(shap_values[1],X_test,plot_type="violin")

shap_interaction_values=explainer.shap_interaction_values(X_test)
shap.summary_plot(shap_interaction_values[1],X_test)

对单个病人
idx=126
patient=X.iloc[idx,:]
patient

shap.summary_plot(shap_interaction_values[1][3],X_test,plot_type='bar')

shap_values_patient=explainer.shap_values(patient)
shap_values_patient

shap_values_patient[1]

shap.force_plot(explainer.expected_value[1],shap_values_patient[1],patient)

瀑布图
展现了某个病人从测试集平均结果到最终预测结果的决策过程,以及各特征对预测结果的贡献影响.
idx=126
patient=X.loc[idx,:]
shap_values_patient=explainer.shap_values(patient)
shap.waterfall_plot(explainer.expected_value[1],shap_values_patient[1],patient)

测试集所有样本的summaryplot
将测试集所有样本的force plot旋转九十度并拼接在一起,形成summary plot
可以在下拉菜单选择按照相似性聚类展示、按照预测结果概率从到小展示,按照测试集原本样本顺序、按照某个特征分别展示。
number_show=60
shap_values_summary=explainer.shap_values(X_train.iloc[:number_show])
shap.force_plot(explainer.expected_value[1],shap_values_summary[1],X_test.iloc[:number_show])

默认是按照相似性显示的,分析相似的病人都具有哪些特征。
Dependence Plot
展示某个特征从小变大时对预测结果的shap值。
shap.dependence_plot("num_major_vessels",shap_values[1],X_test,interaction_index=None)

shap.dependence_plot("max_heart_rate_achieved",shap_values[1],X_test,interaction_index=None)

shap.dependence_plot("max_heart_rate_achieved",shap_values[1],X_test,interaction_index="sex_male")

Partial Dependence Plot
展示某个特征从小变大时模型预测结果.
shap.partial_dependence_plot("max_heart_rate_achieved",model.predict,X_test,model_expected_value=True,feature_expected_value=True)

决策图
瀑布图只能展示单个数据的决策过程,决策图可以展示测试集所有数据的决策过程
决策图主要是展示所有数据的决策路径.
shap.decision_plot(explainer.expected_value[1],shap_values[1],X_test)

查看典型决策路径和异常点
shap.decision_plot(explainer.expected_value[1],shap_values[1],X_test,feature_order="hclust")

shap.decision_plot(explainer.expected_value[1],shap_values[1],X_test,link='logit')

绘制单个样本的决策图
idx=30
selection=np.zeros((61))
selection[idx]=1
selection=selection>0
print("索引号为{}的样本,在原始数据集X中的索引号为{}".format(idx,X_test.iloc[idx:idx+1].index[0]))
shap.decision_plot(explainer.expected_value[1],shap_values[1][selection],X_test[selection])

自定义决策图特征显示顺序
feature_idx=[i for i in range(26)]
idx=30
selection=np.zeros((61))
selection[idx]=1
selection=selection>0
print("索引号为{}的样本,在原始数据集X中的索引号为{}".format(idx,X_test.iloc[idx:idx+1].index[0]))
shap.decision_plot(explainer.expected_value[1],shap_values[1][selection],X_test[selection],feature_order=feature_idx)

选出测试集中模型预测错误的样本
misclassified=y_pred != y_test
misclassified_df=pd.DataFrame({'是否预测错误':misclassified})
misclassified_df
misclassified_df=misclassified_df[misclassified_df['是否预测错误']==True]
misclassified_df
idx=194
patient=X.iloc[idx,:]
patient_df=X.loc[idx:idx]
model_predict_proba=model.predict_proba(patient_df)[0][1]
print('{}号病人的真实标签是{},模型预测为{:.2f}'.format(idx,bool(y_test[idx]),model_predict_proba))
shap_values_patient=explainer.shap_values(patient)
shap.force_plot(explainer.expected_value[1],shap_values_patient[1],patient)

在决策图中显示测试集中模型预测错误的样本
shap.decision_plot(explainer.expected_value[1],shap_values[1],X_test,highlight=misclassified)

shap.decision_plot(explainer.expected_value[1],shap_values[1][misclassified],X_test[misclassified],highlight=range(len(misclassified_df)))

shap.decision_plot(explainer.expected_value[1],
shap_values[1][misclassified],X_test[misclassified],
feature_order="hclust",
highlight=range(len(misclassified_df)))

两两交互特征对预测结果的影响
主对角线的图和summary plot相同
其他的图中,红色表示两个特征的值都较高,蓝色表示两个特征的值都较低。
每个图里,越靠右的点表示这一对两两交互特征对预测为患病的结果有正向影响.
shap_interaction_values=explainer.shap_interaction_values(X_test)
shap.summary_plot(shap_interaction_values[1],X_test)

shap_interaction_values[1][8].shape
import seaborn as sns
plt.figure(figsize=(10,10))
sns.heatmap(shap_interaction_values[1][8],annot=True,fmt='.1f',square=True)
plt.show()

考虑两两交互特征的决策图
shap.decision_plot(explainer.expected_value[1],shap_interaction_values[1],X_test,highlight=misclassified)

shap.decision_plot(explainer.expected_value[1],shap_interaction_values[1],X_test,highlight=misclassified,
feature_display_range=slice(None,None,-1),ignore_warnings=True)

两两交互特征的单个样本决策图
idx=30
selection=np.zeros((61))
selection[idx]=1
selection=selection>0
print("索引号为{}的样本,在原始数据集X中的索引号为{}".format(idx,X_test.iloc[idx:idx+1].index[0]))
shap.decision_plot(explainer.expected_value[1],shap_values[1][selection],X_test[selection])

idx=30
selection=np.zeros((61))
selection[idx]=1
selection=selection>0
print("索引号为{}的样本,在原始数据集X中的索引号为{}".format(idx,X_test.iloc[idx:idx+1].index[0]))
shap.decision_plot(explainer.expected_value[1],shap_values[1][selection],X_test[selection],
feature_display_range=slice(None,None,-1),ignore_warnings=True)

某一个病人的某一特征变化对模型分类结果的影响
idx=25
X_test.loc[idx]

idx=25
shap.decision_plot(explainer.expected_value[1],hypothetical_shap_values[[0,50,99]],X_test.iloc[idx],feature_order="importance")

找出受st_depression特征影响最大的病人
X_test['st_depression'].unique()
idx=np.argpartition(shap_values[1][:,X_test.columns.get_loc('st_depression')],2)
idx=5
patient=X_test.iloc[idx,:]
shap_values_patient=explainer.shap_values(patient)
shap.waterfall_plot(explainer.expected_value[1],shap_values_patient[1],patient)

shap.decision_plot(explainer.expected_value[1],shap_values[1][5],X_test,feature_order="importance")

本文内容由网友自发贡献,版权归原作者所有,本站不承担相应法律责任。如您发现有涉嫌抄袭侵权的内容,请联系:hwhale#tublm.com(使用前将#替换为@)