GOplot
为这个包写笔记,主要是复习一下markdown写作而已,还是建议大家看原作者的英文文档
安装并加载必须的packages
如果你还没有安装,就运行下面的代码安装:
install.packages('GOplot')
library(GOplot)
如果你安装好了,就直接加载它们即可
library(GOplot)
看看内置的测试数据
data(EC)
str(EC)
## List of 5
## $ eset :'data.frame': 20644 obs. of 7 variables:
## ..$ Gene_Symbol: Factor w/ 20644 levels "0610007P14Rik",..: 1 2 3 4 5 6 7 8 9 10 ...
## ..$ Brain_A : num [1:20644] 0.5838 0.1326 -0.0936 0.0671 0.0219 ...
## ..$ Brain_B : num [1:20644] 0.811 0.112 -0.201 0.126 0.135 ...
## ..$ Brain_C : num [1:20644] -0.248 0.312 0.251 -0.123 0.272 ...
## ..$ Heart_A : num [1:20644] -0.671 0.102 -1.021 -0.744 0.275 ...
## ..$ Heart_B : num [1:20644] -0.587 -0.0662 -0.1876 -0.4276 -0.0775 ...
## ..$ Heart_C : num [1:20644] -0.6518 0.0977 -0.6915 -0.4372 0 ...
## $ genelist:'data.frame': 2039 obs. of 7 variables:
## ..$ ID : Factor w/ 2039 levels "0610007P14Rik",..: 1672 1634 496 1673 1591 1651 1105 1639 322 953 ...
## ..$ logFC : num [1:2039] 6.65 6.28 4.48 6.47 5.52 ...
## ..$ AveExpr : num [1:2039] 1.217 1.16 0.837 1.356 2.325 ...
## ..$ t : num [1:2039] 88.7 70 65.6 59.9 58.5 ...
## ..$ P.Value : num [1:2039] 1.32e-18 2.41e-17 5.31e-17 1.62e-16 2.14e-16 ...
## ..$ adj.P.Val: num [1:2039] 2.73e-14 2.49e-13 3.65e-13 8.34e-13 8.81e-13 ...
## ..$ B : num [1:2039] 29 27.6 27.2 26.5 26.3 ...
## $ david :'data.frame': 174 obs. of 5 variables:
## ..$ Category: Factor w/ 3 levels "BP","CC","MF": 1 1 1 1 1 1 1 1 1 1 ...
## ..$ ID : Factor w/ 174 levels "GO:0000165","GO:0000267",..: 54 11 6 155 154 162 127 85 42 40 ...
## ..$ Term : Factor w/ 174 levels "actin binding",..: 69 173 20 169 21 148 153 117 130 141 ...
## ..$ Genes : Factor w/ 148 levels "ACOX1, PPARD, CPT2, ECH1, PTGS2, ABHD5, PTGS1, ACOT1, SC4MOL, FAR1, PRKAR2B, FAR2, ACOT7, TPI1, PTGES, CH25H, ELOVL7, PRKAA2, H"| __truncated__,..: 72 94 95 65 101 49 80 136 137 36 ...
## ..$ adj_pval: num [1:174] 2.17e-06 1.04e-05 7.62e-06 1.19e-04 7.20e-04 ...
## $ genes :'data.frame': 37 obs. of 2 variables:
## ..$ ID : Factor w/ 37 levels "ACVRL1","AMOT",..: 28 16 20 3 9 29 11 27 35 5 ...
## ..$ logFC: num [1:37] -0.653 0.371 2.654 0.87 -2.565 ...
## $ process : chr [1:7] "heart development" "phosphorylation" "vasculature development" "blood vessel development" ...
可以看到测试数据EC里面含有5个数据,其中:
eset是表达矩阵,genelist是差异分析矩阵,
david是GO的超几何分布检验结果,
genes是根据阈值挑选的具有统计学显著的差异基因,
process是挑选的7个GO通路用来可视化。
下面我们可以具体看看david和genelist的数据:
# Get a glimpse of the data format of the results of the functional analysis...
head(EC$david)
## Category ID Term
## 1 BP GO:0007507 heart development
## 2 BP GO:0001944 vasculature development
## 3 BP GO:0001568 blood vessel development
## 4 BP GO:0048729 tissue morphogenesis
## 5 BP GO:0048514 blood vessel morphogenesis
## 6 BP GO:0051336 regulation of hydrolase activity
## Genes
## 1 DLC1, NRP2, NRP1, EDN1, PDLIM3, GJA1, TTN, GJA5, ZIC3, TGFB2, CERKL, GATA6, COL4A3BP, GAB1, SEMA3C, MKL2, SLC22A5, MB, PTPRJ, RXRA, VANGL2, MYH6, TNNT2, HHEX, MURC, MIB1, FOXC2, FOXC1, ADAM19, MYL2, TCAP, EGLN1, SOX9, ITGB1, CHD7, HEXIM1, PKD2, NFATC4, PCSK5, ACTC1, TGFBR2, NF1, HSPG2, SMAD3, TBX1, TNNI3, CSRP3, FOXP1, KCNJ8, PLN, TSC2, ATP6V0A1, TGFBR3, HDAC9
## 2 GNA13, ACVRL1, NRP1, PGF, IL18, LEPR, EDN1, GJA1, FOXO1, GJA5, TGFB2, WARS, CERKL, APOE, CXCR4, ANG, SEMA3C, NOS2, MKL2, FGF2, RAPGEF1, PTPRJ, RECK, EFNB2, VASH1, PNPLA6, THY1, MIB1, NUS1, FOXC2, FOXC1, CAV1, CDH2, MEIS1, WT1, CDH5, PTK2, FBXW8, CHD7, PLCD1, PLXND1, FIGF, PPAP2B, MAP2K1, TBX4, TGFBR2, NF1, TBX1, TNNI3, LAMA4, MEOX2, ECSCR, HBEGF, AMOT, TGFBR3, HDAC7
## 3 GNA13, ACVRL1, NRP1, PGF, IL18, LEPR, EDN1, GJA1, FOXO1, GJA5, TGFB2, WARS, CERKL, APOE, CXCR4, ANG, SEMA3C, NOS2, MKL2, FGF2, RAPGEF1, PTPRJ, RECK, VASH1, PNPLA6, THY1, MIB1, NUS1, FOXC2, FOXC1, CAV1, CDH2, MEIS1, WT1, CDH5, PTK2, FBXW8, CHD7, PLCD1, PLXND1, FIGF, PPAP2B, MAP2K1, TBX4, TGFBR2, NF1, TBX1, TNNI3, LAMA4, MEOX2, ECSCR, HBEGF, AMOT, TGFBR3, HDAC7
## 4 DLC1, ENAH, NRP1, PGF, ZIC2, TGFB2, CD44, ILK, SEMA3C, RET, AR, RXRA, VANGL2, LEF1, TNNT2, HHEX, MIB1, NCOA3, FOXC2, FOXC1, TGFB1I1, WNT5A, COBL, BBS4, FGFR3, TNC, BMPR2, CTNND1, EGLN1, NR3C1, SOX9, TCF7L1, IGF1R, FOXQ1, MACF1, HOXA5, BCL2, PLXND1, CAR2, ACTC1, TBX4, SMAD3, FZD3, SHANK3, FZD6, HOXB4, FREM2, TSC2, ZIC5, TGFBR3, APAF1
## 5 GNA13, CAV1, ACVRL1, NRP1, PGF, IL18, LEPR, EDN1, GJA1, CDH2, MEIS1, WT1, TGFB2, WARS, PTK2, CERKL, APOE, CXCR4, ANG, SEMA3C, PLCD1, NOS2, MKL2, PLXND1, FIGF, FGF2, PTPRJ, TGFBR2, TBX4, NF1, TBX1, TNNI3, PNPLA6, VASH1, THY1, NUS1, MEOX2, ECSCR, AMOT, HBEGF, FOXC2, FOXC1, HDAC7
## 6 CAV1, XIAP, AGFG1, ADORA2A, TNNC1, TBC1D9, LEPR, ABHD5, EDN1, ASAP2, ASAP3, SMAP1, TBC1D12, ANG, TBC1D14, MTCH1, TBC1D13, TBC1D4, TBC1D30, DHCR24, HIP1, VAV3, NOS1, NF1, MYH6, RICTOR, TBC1D22A, THY1, PLCE1, RNF7, NDEL1, CHML, IFT57, ACAP2, TSC2, ERN1, APAF1, ARAP3, ARAP2, ARAP1, HTR2A, F2R
## adj_pval
## 1 0.000002170
## 2 0.000010400
## 3 0.000007620
## 4 0.000119000
## 5 0.000720000
## 6 0.001171166
# ...and of the data frame of selected genes
head(EC$genelist)
## ID logFC AveExpr t P.Value adj.P.Val B
## 1 Slco1a4 6.645388 1.2168670 88.65515 1.32e-18 2.73e-14 29.02715
## 2 Slc19a3 6.281525 1.1600468 69.95094 2.41e-17 2.49e-13 27.62917
## 3 Ddc 4.483338 0.8365231 65.57836 5.31e-17 3.65e-13 27.18476
## 4 Slco1c1 6.469384 1.3558865 59.87613 1.62e-16 8.34e-13 26.51242
## 5 Sema3c 5.515630 2.3252117 58.53141 2.14e-16 8.81e-13 26.33626
## 6 Slc38a3 4.761755 0.9218670 54.11559 5.58e-16 1.76e-12 25.70308
Generate the plotting object
构造绘图对象
整个包都基于这个对象在进行各种可视化
circ <- circle_dat(EC$david, EC$genelist)
head(circ)
## category ID term count genes logFC adj_pval
## 1 BP GO:0007507 heart development 54 DLC1 -0.9707875 2.17e-06
## 2 BP GO:0007507 heart development 54 NRP2 -1.5153173 2.17e-06
## 3 BP GO:0007507 heart development 54 NRP1 -1.1412315 2.17e-06
## 4 BP GO:0007507 heart development 54 EDN1 1.3813006 2.17e-06
## 5 BP GO:0007507 heart development 54 PDLIM3 -0.8876939 2.17e-06
## 6 BP GO:0007507 heart development 54 GJA1 -0.8179480 2.17e-06
## zscore
## 1 -0.8164966
## 2 -0.8164966
## 3 -0.8164966
## 4 -0.8164966
## 5 -0.8164966
## 6 -0.8164966
当然,你可以用str等函数具体看看这个对象里面到底有什么,共8列信息:
category
ID
term
count
gene
logFC
adj_pval
zscore
下面就是这个包的各种可视化函数了,都是针对于上面circle_dat函数构造好的绘图对象来的,就是一些绘图参数需要调整而已,没什么需要做笔记及认真讲解的,所有的重点都是测试数据里面的5个对象是如何来的,就是表达矩阵的差异分析而已。
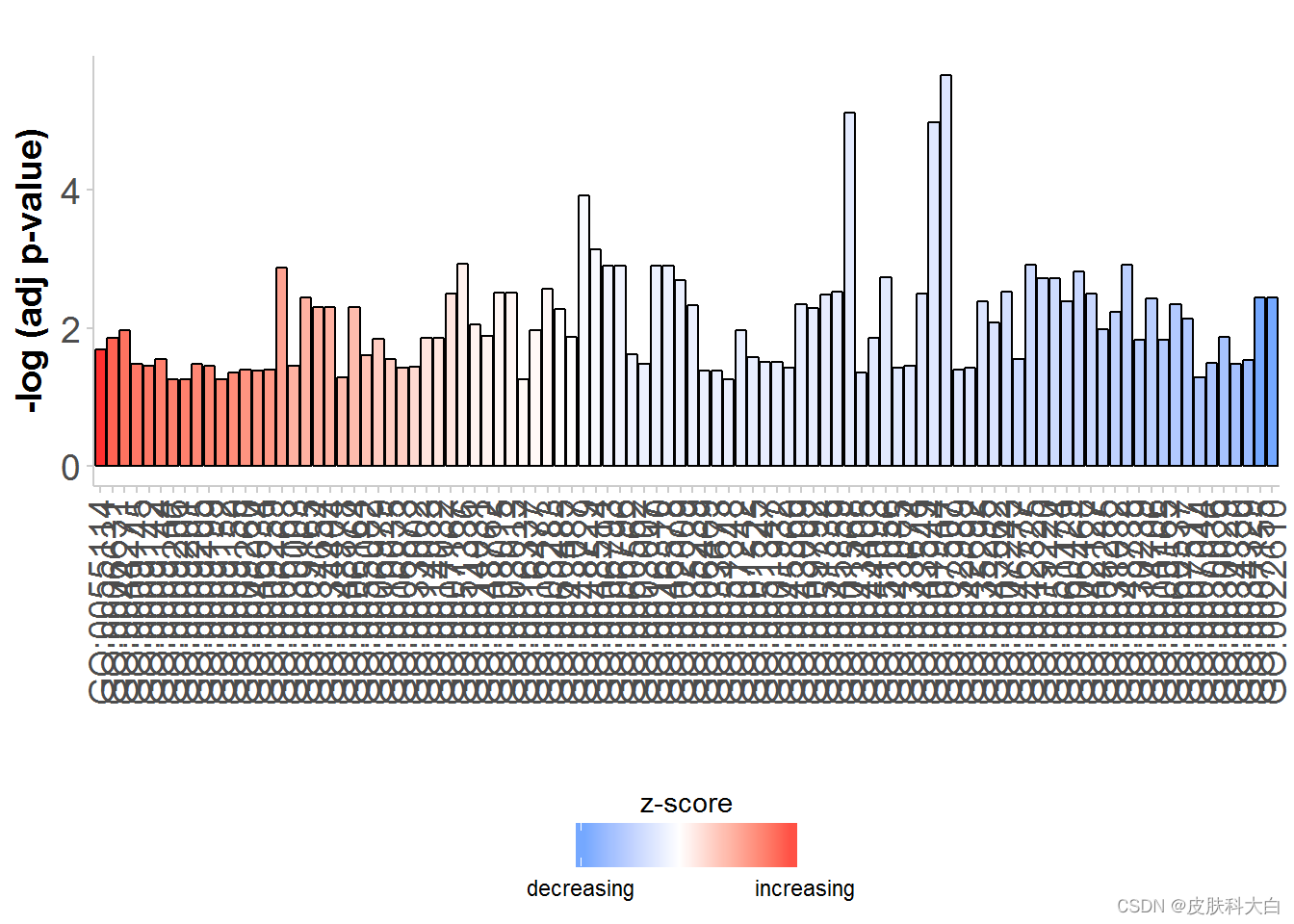
# Generate a simple barplot
GOBar(subset(circ, category == 'BP'))
# Facet the barplot according to the categories of the terms
GOBar(circ, display = 'multiple')
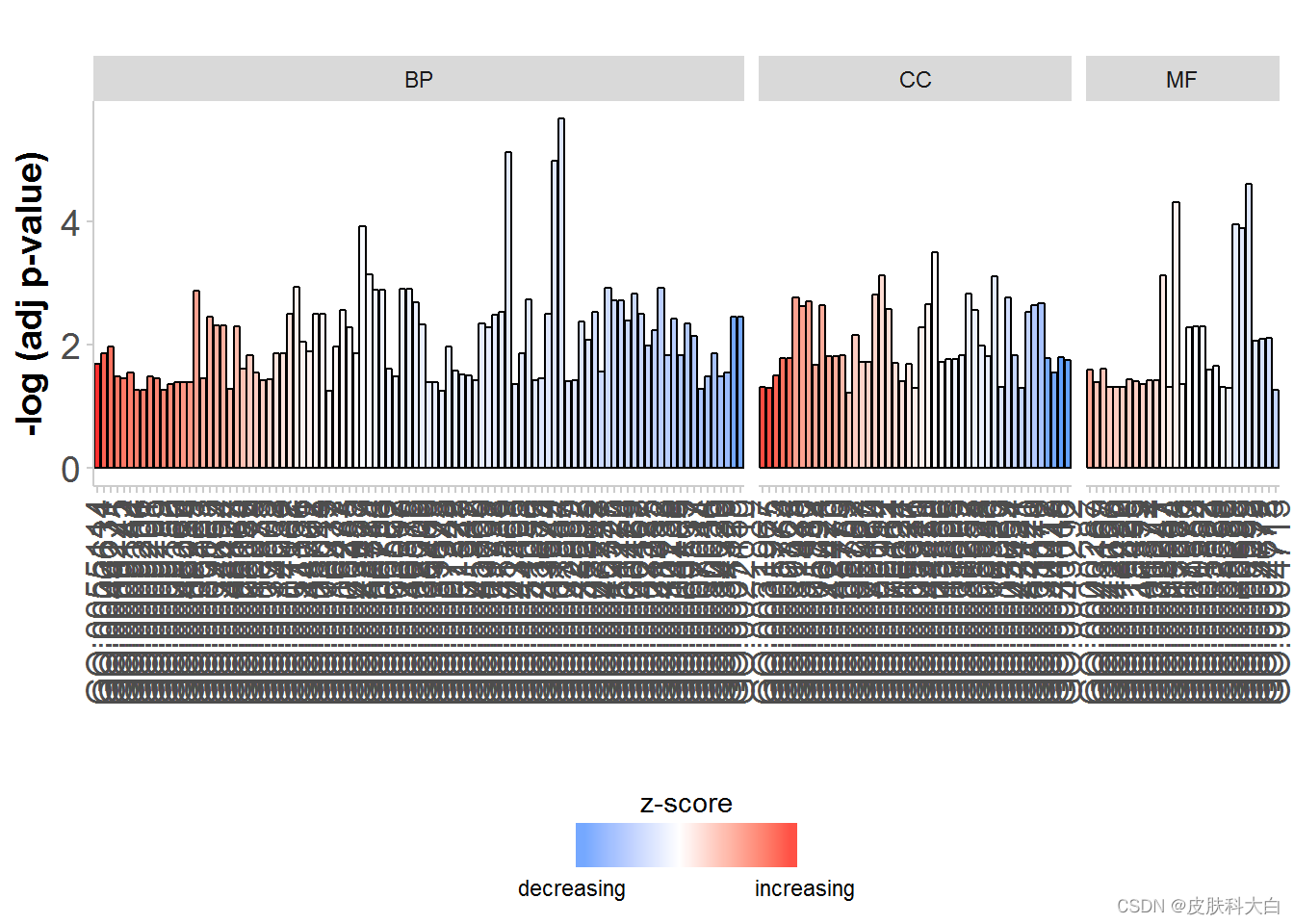
# Facet the barplot, add a title and change the colour scale for the z-score
GOBar(circ, display = 'multiple', title = 'Z-score coloured barplot', zsc.col = c('yellow', 'black', 'cyan'))

# Generate the bubble plot with a label threshold of 3
GOBubble(circ, labels = 3)
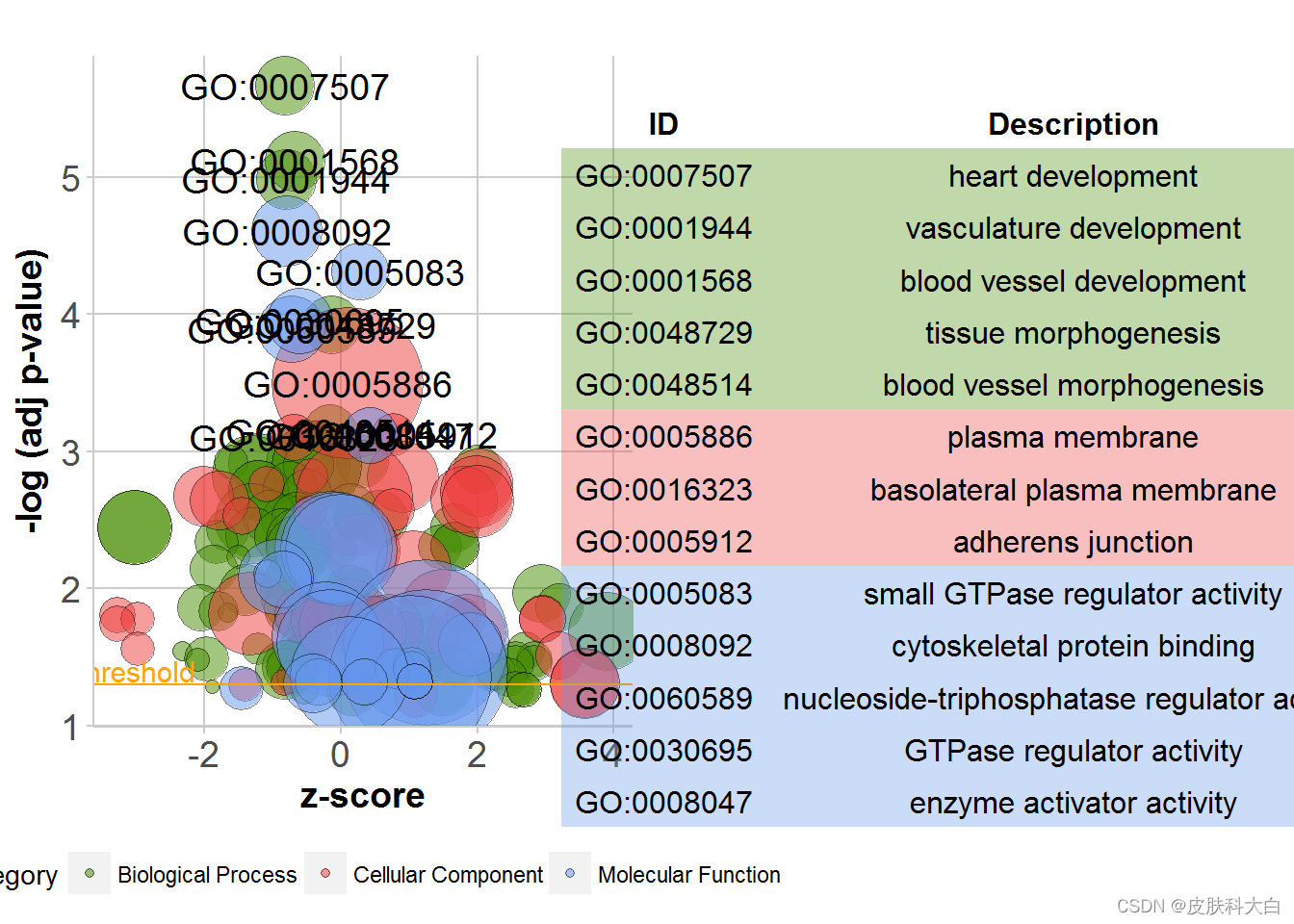
# Add a title, change the colour of the circles, facet the plot according to the categories and change the label threshold
GOBubble(circ, title = 'Bubble plot', colour = c('orange', 'darkred', 'gold'), display = 'multiple', labels = 3)
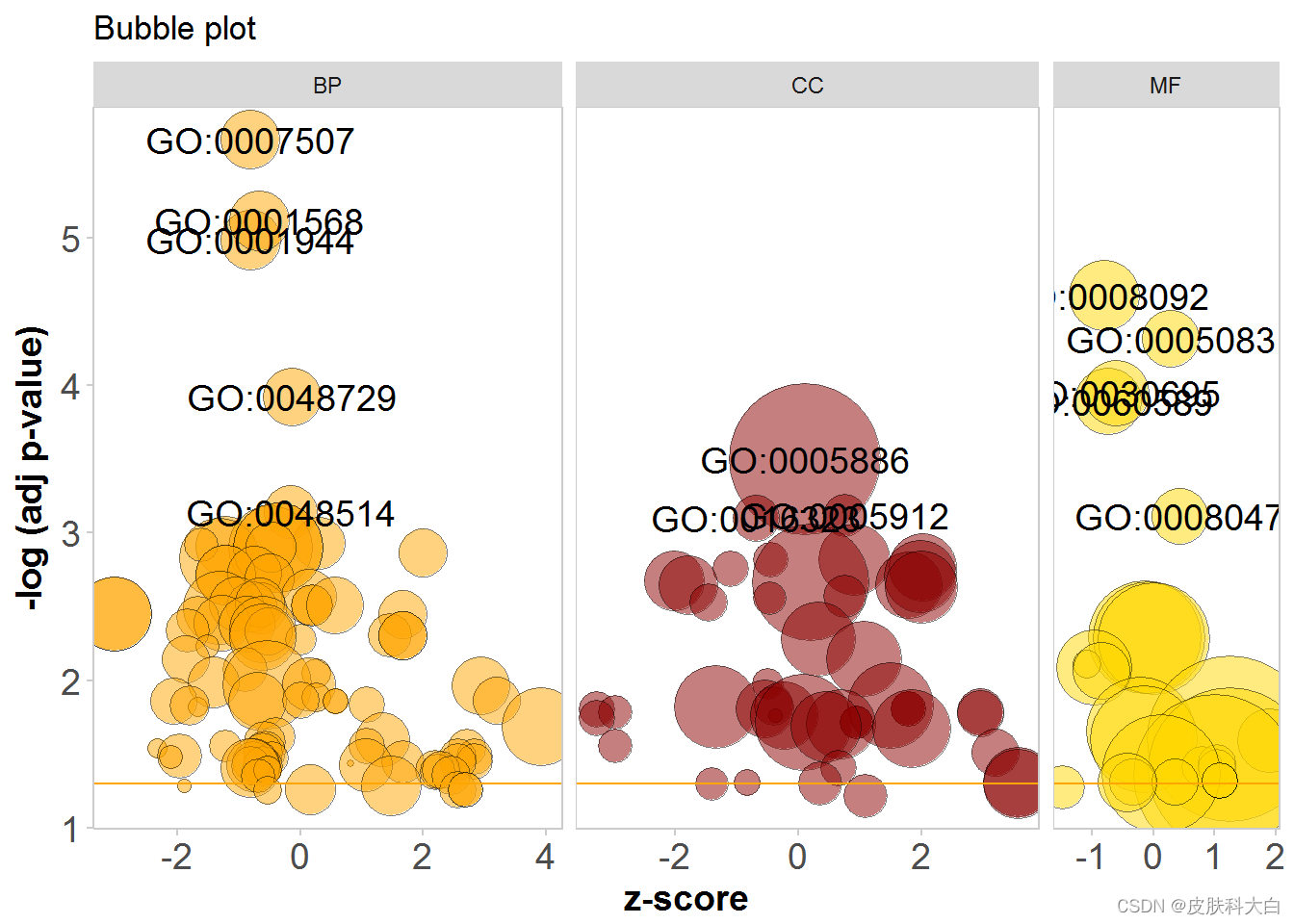
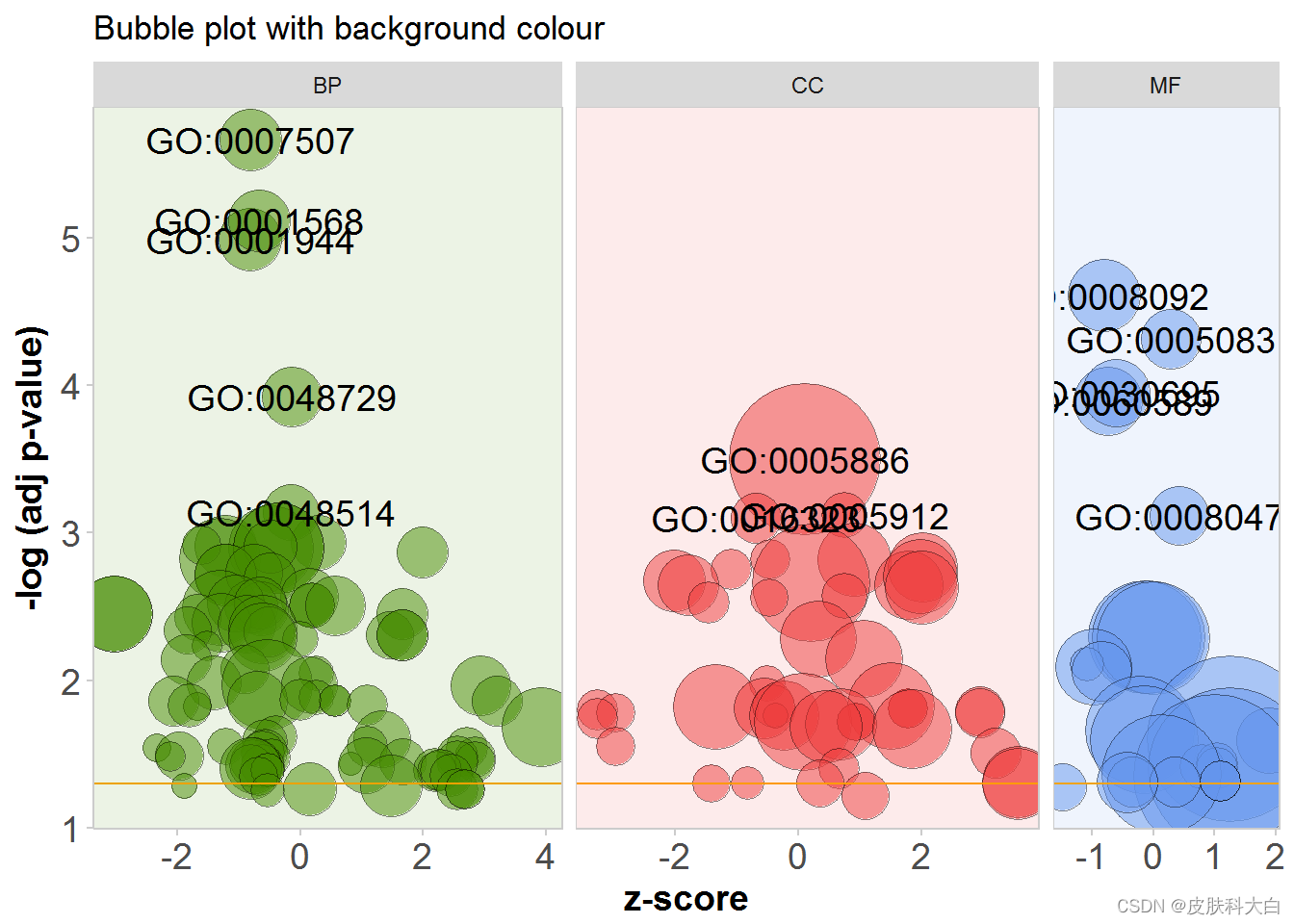
# Colour the background according to the category
GOBubble(circ, title = 'Bubble plot with background colour', display = 'multiple', bg.col = T, labels = 3)
# Reduce redundant terms with a gene overlap >= 0.75...
reduced_circ <- reduce_overlap(circ, overlap = 0.75)
# ...and plot it
GOBubble(reduced_circ, labels = 2.8)

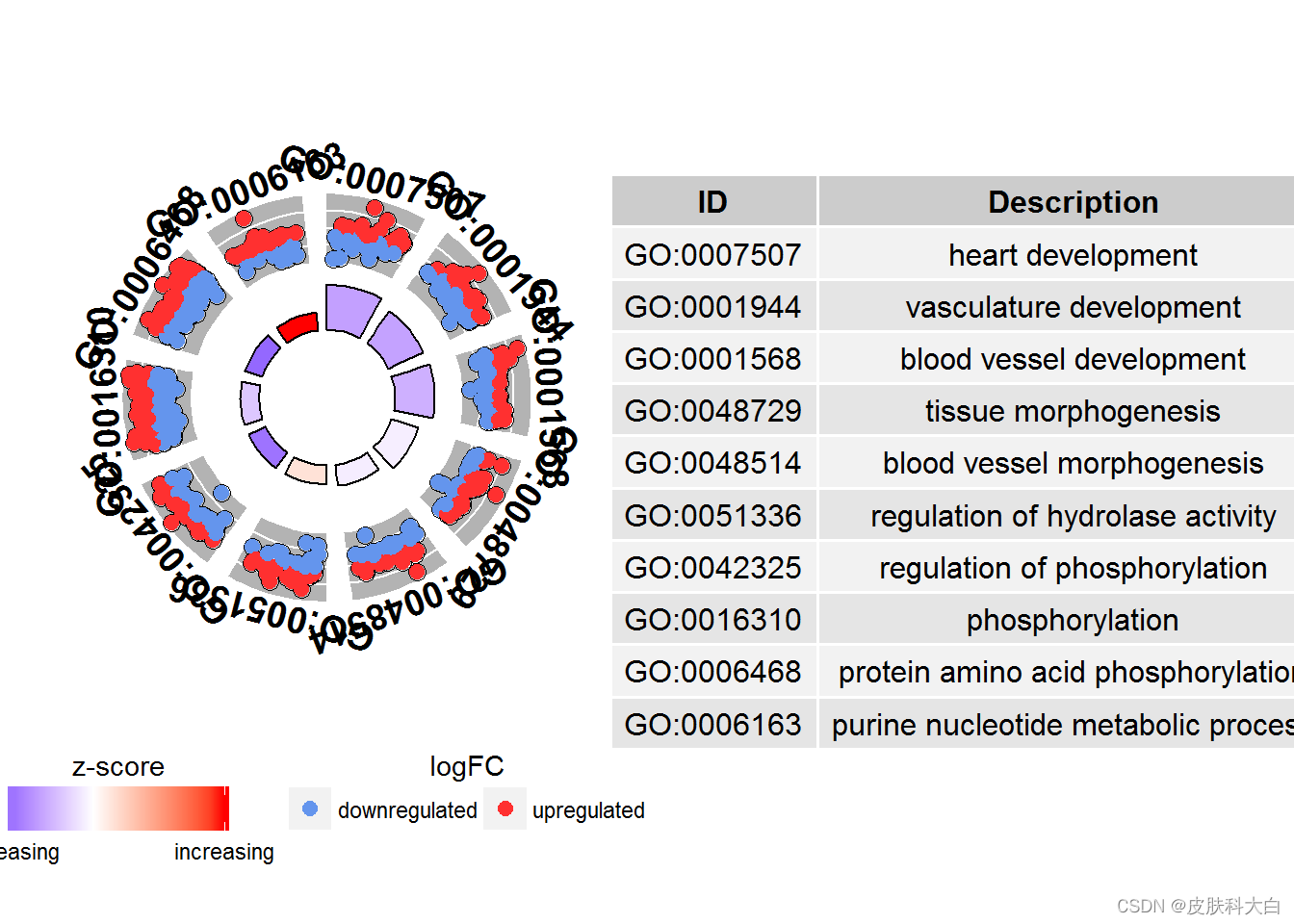
#Generate a circular visualization of the results of gene- annotation enrichment analysis
GOCircle(circ)
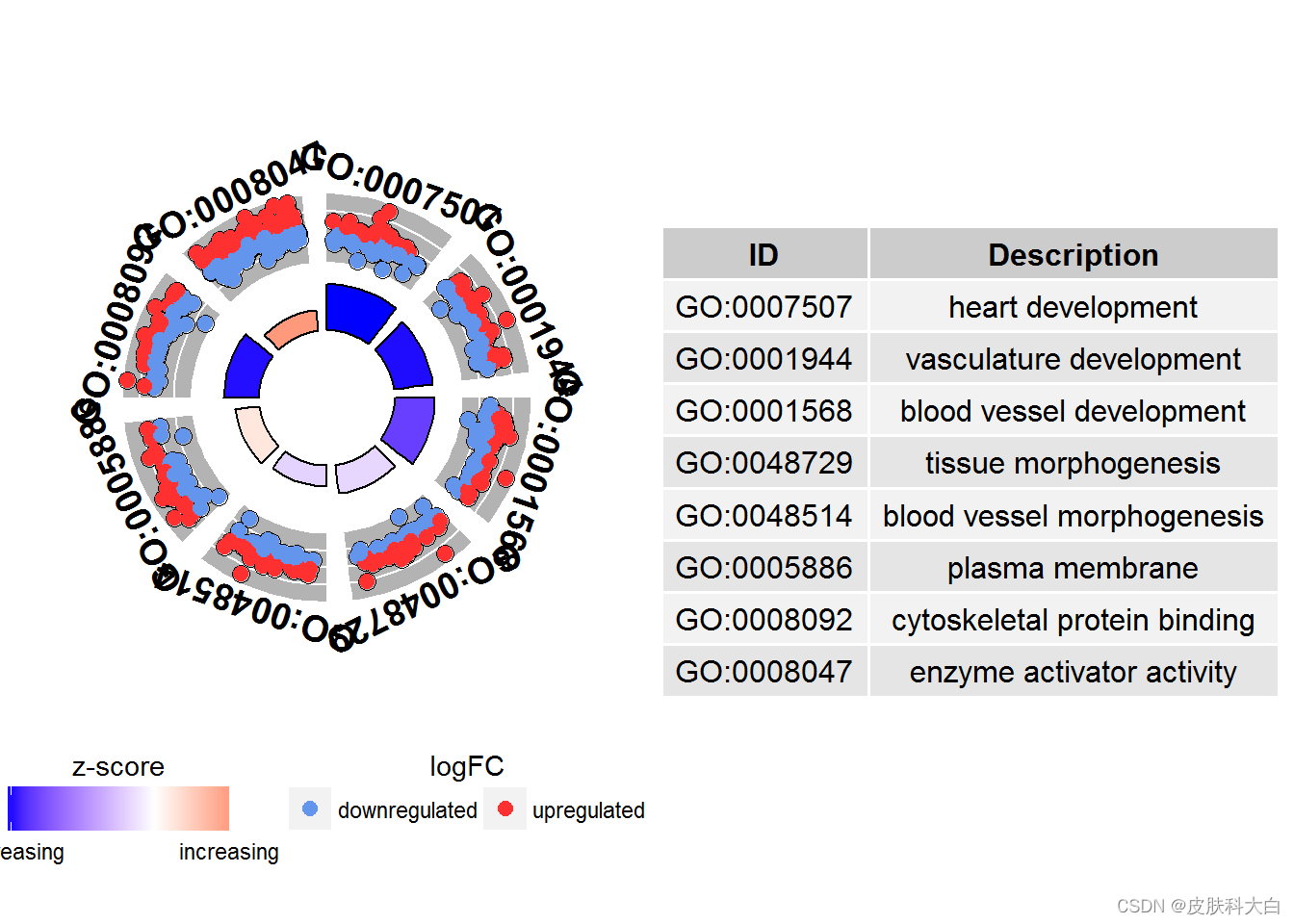
# Generate a circular visualization of selected terms
IDs <- c('GO:0007507', 'GO:0001568', 'GO:0001944', 'GO:0048729', 'GO:0048514', 'GO:0005886', 'GO:0008092', 'GO:0008047')
GOCircle(circ, nsub = IDs)
# Define a list of genes which you think are interesting to look at. The item EC$genes of the toy
# sample contains the data frame of selected genes and their logFC. Have a look...
head(EC$genes)
## ID logFC
## 1 PTK2 -0.6527904
## 2 GNA13 0.3711599
## 3 LEPR 2.6539788
## 4 APOE 0.8698346
## 5 CXCR4 -2.5647537
## 6 RECK 3.6926860
# Since we have a lot of significantly enriched processes we selected some specific ones (EC$process)
EC$process
## [1] "heart development" "phosphorylation"
## [3] "vasculature development" "blood vessel development"
## [5] "tissue morphogenesis" "cell adhesion"
## [7] "plasma membrane"
# Now it is time to generate the binary matrix
chord <- chord_dat(circ, EC$genes, EC$process)
head(chord)
## heart development phosphorylation vasculature development
## PTK2 0 1 1
## GNA13 0 0 1
## LEPR 0 0 1
## APOE 0 0 1
## CXCR4 0 0 1
## RECK 0 0 1
## blood vessel development tissue morphogenesis cell adhesion
## PTK2 1 0 0
## GNA13 1 0 0
## LEPR 1 0 0
## APOE 1 0 0
## CXCR4 1 0 0
## RECK 1 0 0
## plasma membrane logFC
## PTK2 1 -0.6527904
## GNA13 1 0.3711599
## LEPR 1 2.6539788
## APOE 1 0.8698346
## CXCR4 1 -2.5647537
## RECK 1 3.6926860
# Generate the matrix with a list of selected genes
chord <- chord_dat(data = circ, genes = EC$genes)
# Generate the matrix with selected processes
chord <- chord_dat(data = circ, process = EC$process)
# Create the plot
chord <- chord_dat(data = circ, genes = EC$genes, process = EC$process)
GOChord(chord, space = 0.02, gene.order = 'logFC', gene.space = 0.25, gene.size = 5)
## Warning: Using size for a discrete variable is not advised.
## Warning: Removed 7 rows containing missing values (geom_point).
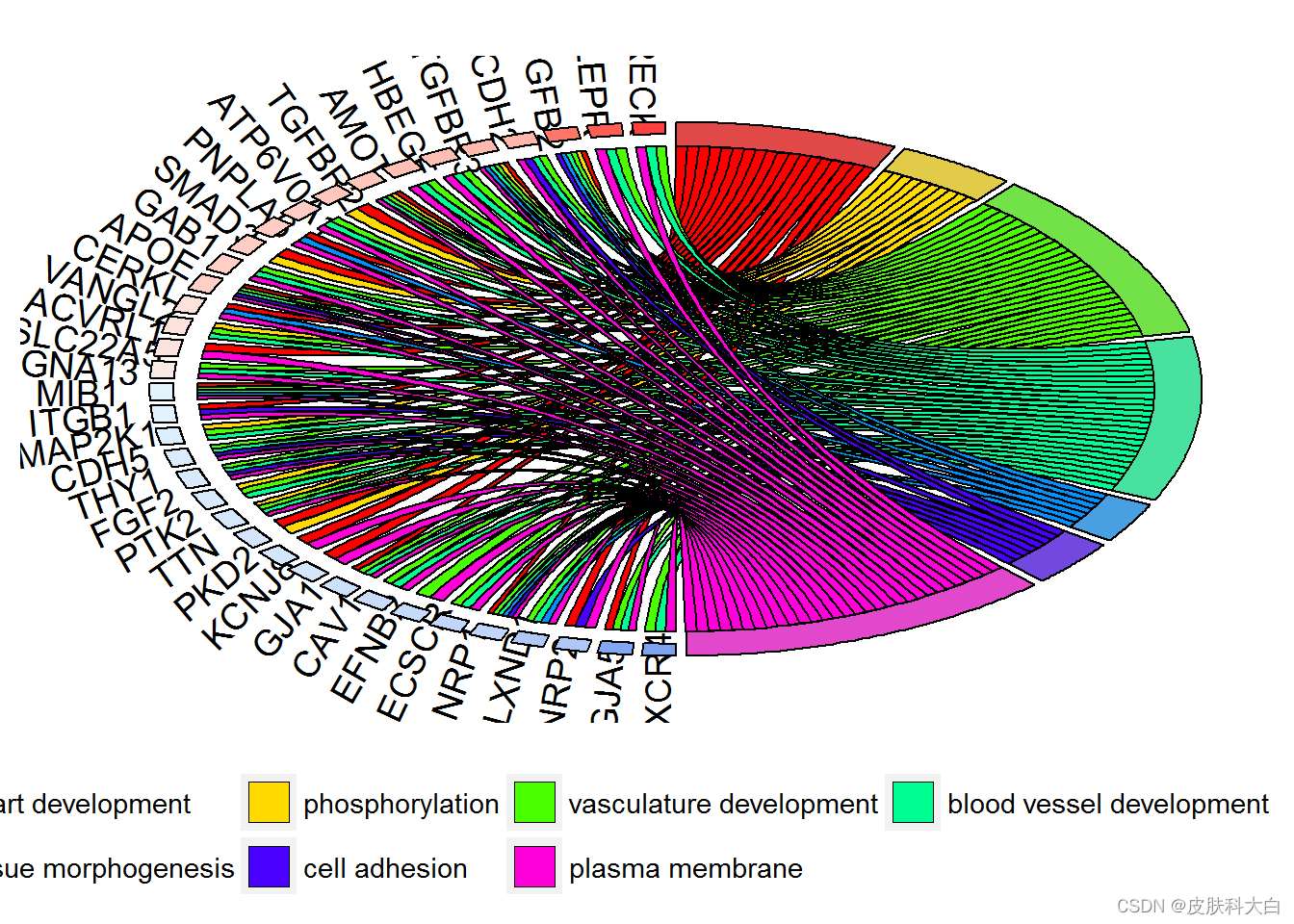
# Display only genes which are assigned to at least three processes
GOChord(chord, limit = c(3, 0), gene.order = 'logFC')
## Warning: Using size for a discrete variable is not advised.
## Warning: Removed 7 rows containing missing values (geom_point).

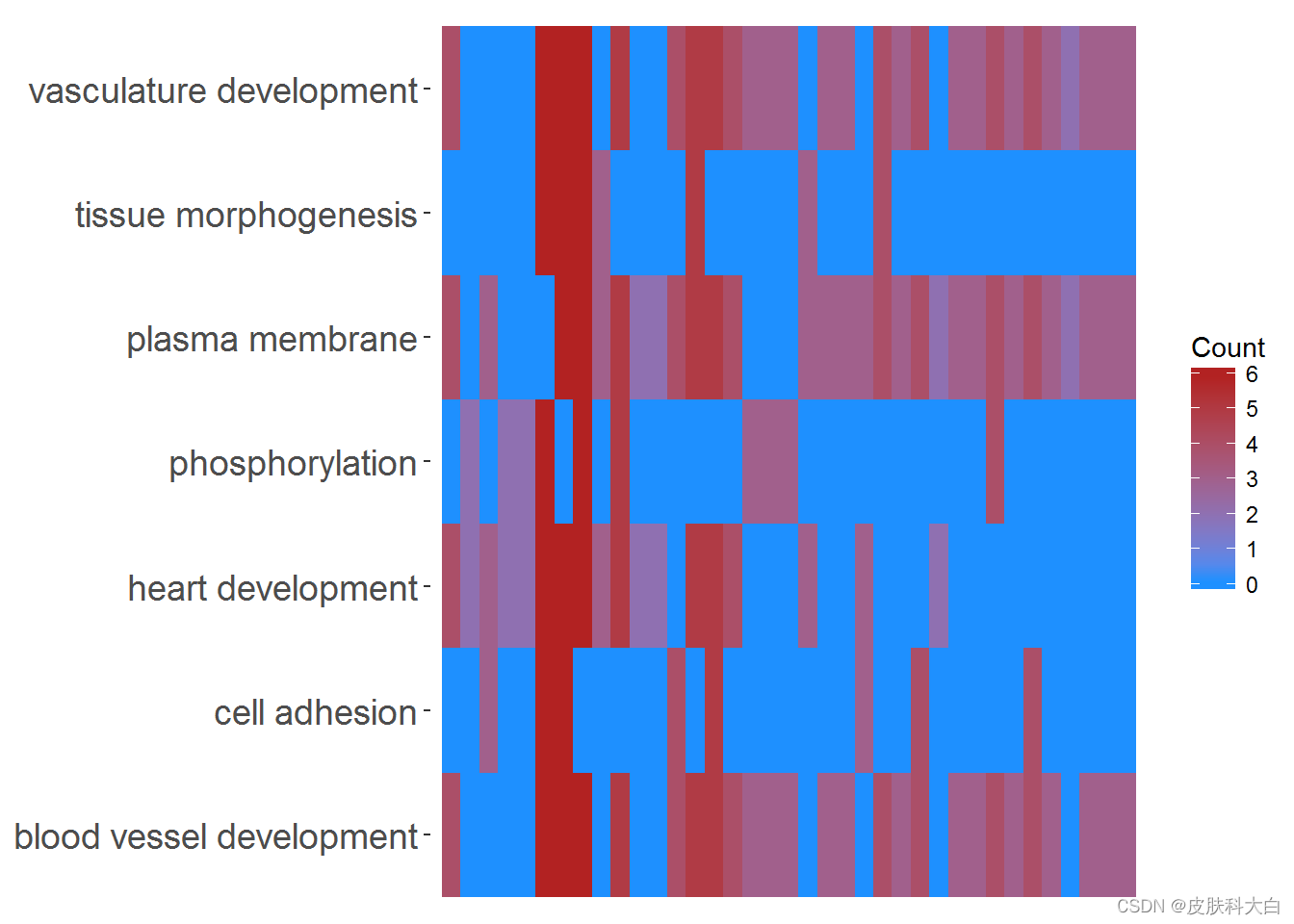
# First, we use the chord object without logFC column to create the heatmap
GOHeat(chord[,-8], nlfc = 0)
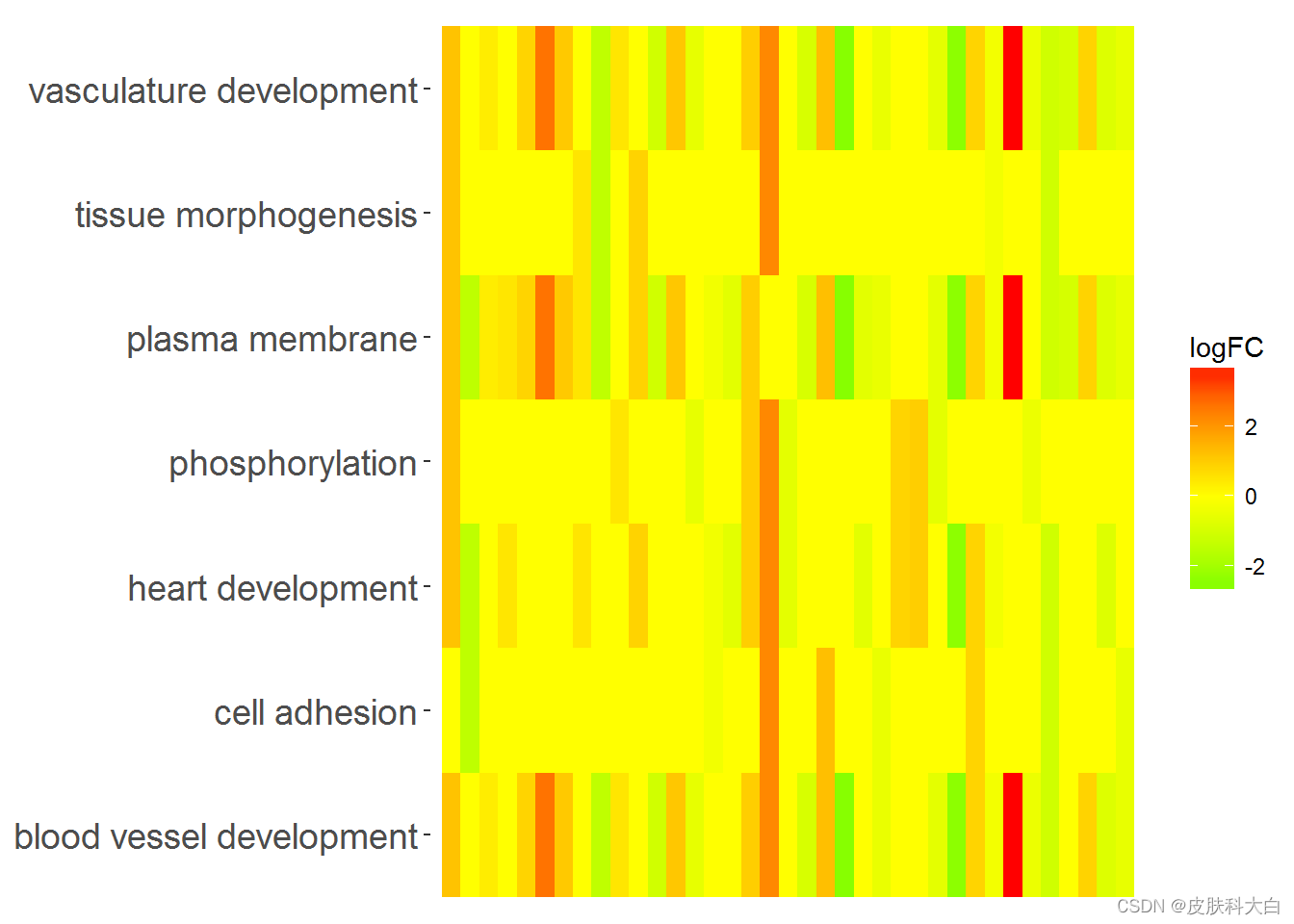
# Now we create the heatmap with logFC values and user-defined colour scale
GOHeat(chord, nlfc = 1, fill.col = c('red', 'yellow', 'green'))
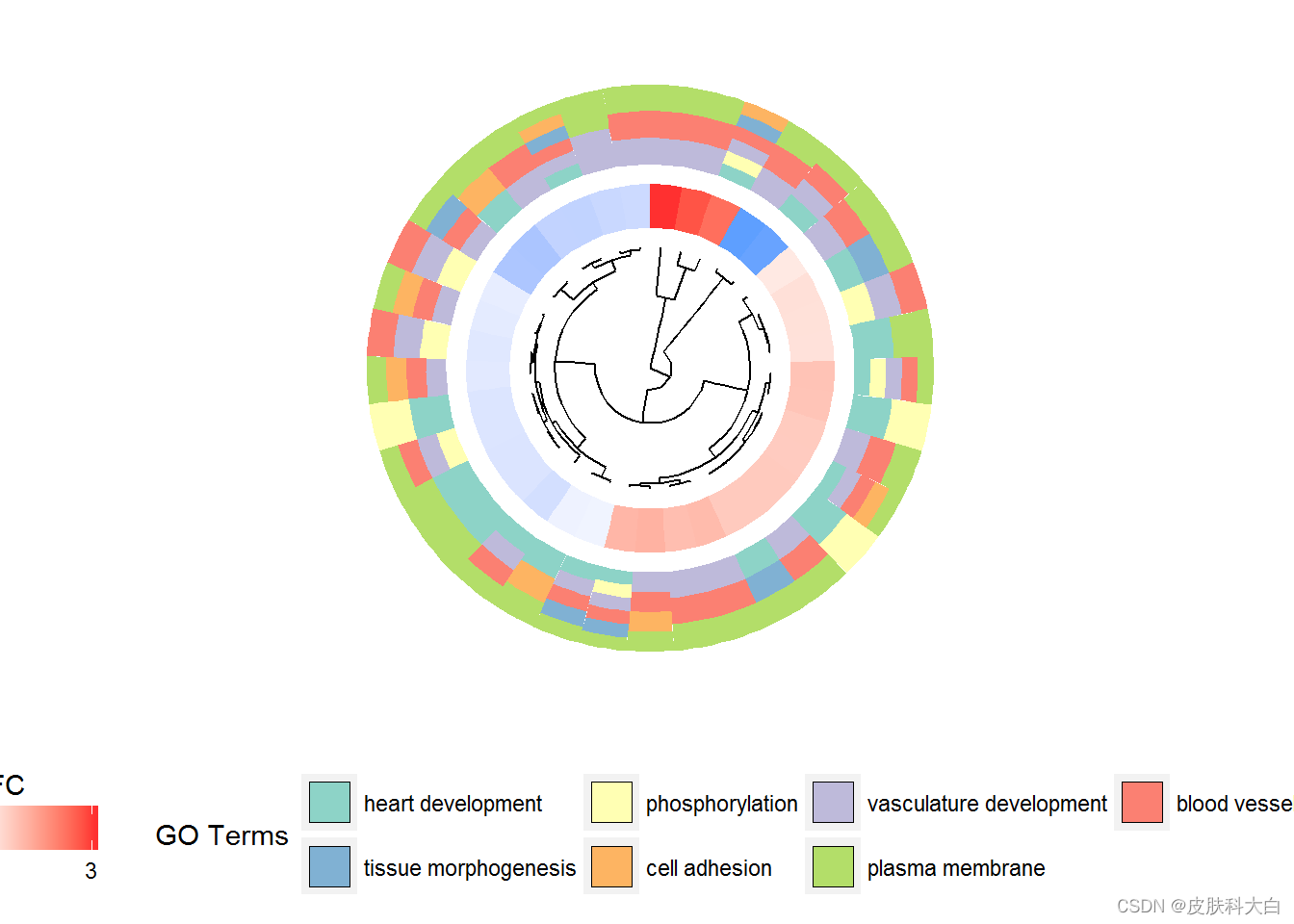
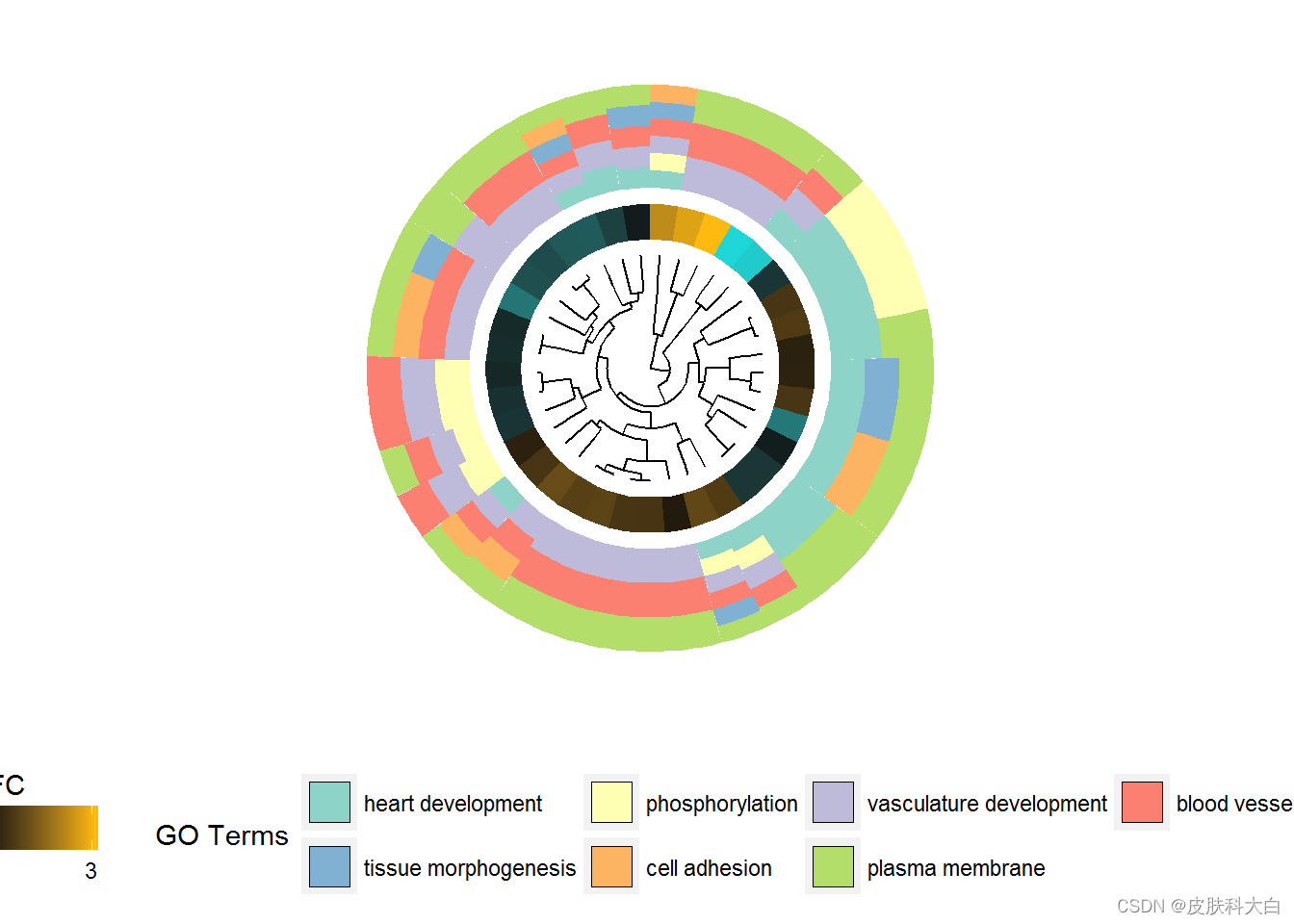
GOCluster(circ, EC$process, clust.by = 'logFC', term.width = 2)
## Warning: Using size for a discrete variable is not advised.
## Warning: Removed 7 rows containing missing values (geom_point).
GOCluster(circ, EC$process, clust.by = 'term', lfc.col = c('darkgoldenrod1', 'black', 'cyan1'))
## Warning: Using size for a discrete variable is not advised.
## Warning: Removed 7 rows containing missing values (geom_point).
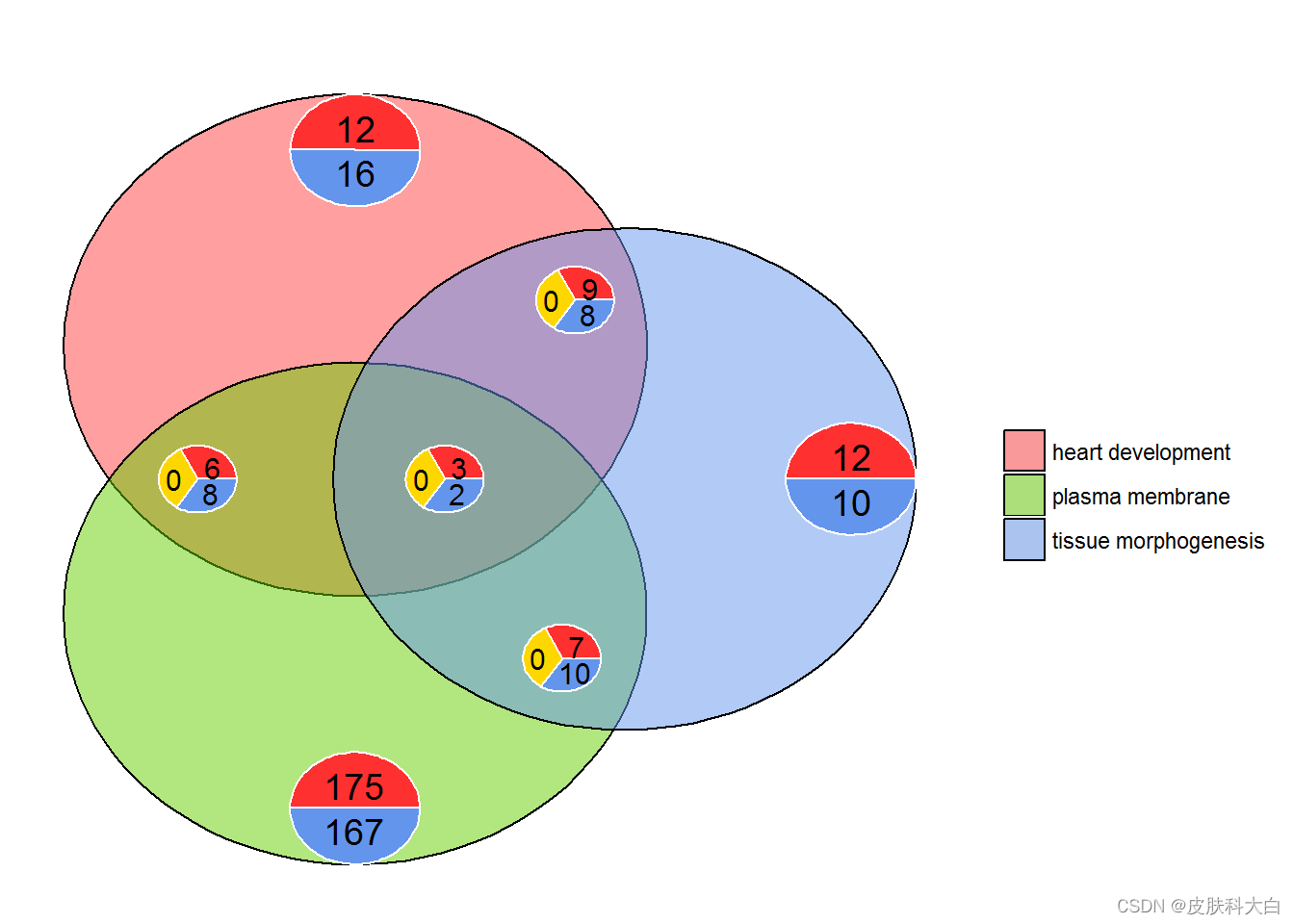
l1 <- subset(circ, term == 'heart development', c(genes,logFC))
l2 <- subset(circ, term == 'plasma membrane', c(genes,logFC))
l3 <- subset(circ, term == 'tissue morphogenesis', c(genes,logFC))
GOVenn(l1,l2,l3, label = c('heart development', 'plasma membrane', 'tissue morphogenesis'))