用deconstructSigs来做cosmic的mutation signature图
作者的英文文档对这个包的用法描述的非常清楚, 我只是记录一下自己学习该包用法的一点感悟。
安装并加载必须的packages
如果你还没有安装,就运行下面的代码安装:
source('http://bioconductor.org/biocLite.R');
install.packages('deconstructSigs')
# dependencies 'BSgenome', 'BSgenome.Hsapiens.UCSC.hg19'
BiocInstaller::biocLite('BSgenome')
BiocInstaller::biocLite('BSgenome.Hsapiens.UCSC.hg19')
如果你安装好了,就直接加载它们即可
suppressPackageStartupMessages(library("deconstructSigs"))
输入数据的准备
head(sample.mut.ref)
## Sample chr pos ref alt
## 1 1 chr1 905907 A T
## 2 1 chr1 1192480 C A
## 3 1 chr1 1854885 G C
## 4 1 chr1 9713992 G A
## 5 1 chr1 12908093 C A
## 6 1 chr1 17257855 C T
class(sample.mut.ref)
## [1] "data.frame"
dim(sample.mut.ref)
## [1] 657 5
只需要一个很简单的数据框即可,5列,分别是样本ID,chr,pos,ref,alt ,很容易从MAF文件里面提取。
理解包自带的signature数据
head(t(signatures.nature2013))
## Signature.1A Signature.1B Signature.2 Signature.3 Signature.4
## A[C>A]A 0.0112 0.0104 0.0105 0.0240 0.0365
## A[C>A]C 0.0092 0.0093 0.0061 0.0197 0.0309
## A[C>A]G 0.0015 0.0016 0.0013 0.0019 0.0183
## A[C>A]T 0.0063 0.0067 0.0037 0.0172 0.0243
## C[C>A]A 0.0067 0.0090 0.0061 0.0194 0.0461
## C[C>A]C 0.0074 0.0047 0.0012 0.0161 0.0614
## Signature.5 Signature.6 Signature.7 Signature.8 Signature.9
## A[C>A]A 0.0149 0.0017 0.0004 0.0368 0.0120
## A[C>A]C 0.0089 0.0028 0.0005 0.0287 0.0067
## A[C>A]G 0.0022 0.0005 0.0000 0.0017 0.0005
## A[C>A]T 0.0092 0.0019 0.0004 0.0300 0.0068
## C[C>A]A 0.0097 0.0101 0.0012 0.0303 0.0098
## C[C>A]C 0.0050 0.0241 0.0006 0.0203 0.0057
## Signature.10 Signature.11 Signature.12 Signature.13 Signature.14
## A[C>A]A 0.0007 0.0002 0.0077 0.0007 0.0001
## A[C>A]C 0.0010 0.0010 0.0047 0.0001 0.0042
## A[C>A]G 0.0003 0.0000 0.0017 0.0001 0.0005
## A[C>A]T 0.0092 0.0002 0.0046 0.0002 0.0296
## C[C>A]A 0.0031 0.0007 0.0135 0.0035 0.0056
## C[C>A]C 0.0009 0.0017 0.0112 0.0011 0.0102
## Signature.15 Signature.16 Signature.17 Signature.18 Signature.19
## A[C>A]A 0.0013 0.0161 0.0018 0.0500 0.0107
## A[C>A]C 0.0040 0.0097 0.0003 0.0076 0.0074
## A[C>A]G 0.0000 0.0022 0.0000 0.0017 0.0005
## A[C>A]T 0.0057 0.0088 0.0032 0.0181 0.0074
## C[C>A]A 0.0106 0.0159 0.0010 0.0965 0.0112
## C[C>A]C 0.0084 0.0100 0.0004 0.0219 0.0159
## Signature.20 Signature.21 Signature.R1 Signature.R2 Signature.R3
## A[C>A]A 0.0013 0.0001 0.0210 0.0137 0.0044
## A[C>A]C 0.0024 0.0007 0.0065 0.0046 0.0047
## A[C>A]G 0.0000 0.0000 0.0000 0.0048 0.0003
## A[C>A]T 0.0029 0.0006 0.0058 0.0081 0.0034
## C[C>A]A 0.0178 0.0020 0.0076 0.3117 0.0156
## C[C>A]C 0.0375 0.0014 0.0023 0.1477 0.0072
## Signature.U1 Signature.U2
## A[C>A]A 0.0105 0.0221
## A[C>A]C 0.0005 0.0123
## A[C>A]G 0.0000 0.0028
## A[C>A]T 0.0112 0.0118
## C[C>A]A 0.0173 0.0057
## C[C>A]C 0.0051 0.0081
head(t(signatures.cosmic))
## Signature.1 Signature.2 Signature.3 Signature.4 Signature.5
## A[C>A]A 0.011098326 6.827082e-04 0.02217231 0.0365 0.014941548
## A[C>A]C 0.009149341 6.191072e-04 0.01787168 0.0309 0.008960918
## A[C>A]G 0.001490070 9.927896e-05 0.00213834 0.0183 0.002207846
## A[C>A]T 0.006233885 3.238914e-04 0.01626515 0.0243 0.009206905
## C[C>A]A 0.006595870 6.774450e-04 0.01878173 0.0461 0.009674904
## C[C>A]C 0.007342368 2.136810e-04 0.01576046 0.0614 0.004952301
## Signature.6 Signature.7 Signature.8 Signature.9 Signature.10
## A[C>A]A 0.0017 0.0004 0.036718004 0.0120 0.0007
## A[C>A]C 0.0028 0.0005 0.033245722 0.0067 0.0010
## A[C>A]G 0.0005 0.0000 0.002525311 0.0005 0.0003
## A[C>A]T 0.0019 0.0004 0.033598550 0.0068 0.0092
## C[C>A]A 0.0101 0.0012 0.031723757 0.0098 0.0031
## C[C>A]C 0.0241 0.0006 0.025505407 0.0057 0.0009
## Signature.11 Signature.12 Signature.13 Signature.14 Signature.15
## A[C>A]A 0.0002 0.0077 3.347572e-04 0.0001 0.0013
## A[C>A]C 0.0010 0.0047 6.487361e-04 0.0042 0.0040
## A[C>A]G 0.0000 0.0017 3.814459e-05 0.0005 0.0000
## A[C>A]T 0.0002 0.0046 8.466585e-04 0.0296 0.0057
## C[C>A]A 0.0007 0.0135 1.710090e-03 0.0056 0.0106
## C[C>A]C 0.0017 0.0112 1.159257e-03 0.0102 0.0084
## Signature.16 Signature.17 Signature.18 Signature.19 Signature.20
## A[C>A]A 0.0161 1.832019e-03 0.050536419 0.0107 1.179962e-03
## A[C>A]C 0.0097 3.422356e-04 0.010939825 0.0074 2.211505e-03
## A[C>A]G 0.0022 1.576225e-06 0.002288073 0.0005 1.616910e-07
## A[C>A]T 0.0088 3.179665e-03 0.019424091 0.0074 3.008010e-03
## C[C>A]A 0.0159 1.032430e-03 0.088768109 0.0112 1.737711e-02
## C[C>A]C 0.0100 4.218801e-04 0.020641391 0.0159 3.650246e-02
## Signature.21 Signature.22 Signature.23 Signature.24 Signature.25
## A[C>A]A 0.0001 0.0015040704
```bash
0.02864599 0.009896768
## A[C>A]C 0.0007 0.0024510110 0.0003668005 0.02021464 0.006998929
## A[C>A]G 0.0000 0.0000000000
0.0000000000
0.02047900 0.001448443
A[C>A]T 0.0006 0.0009224525 0.0000000000
0.02460015 0.004966565
C[C>A]A 0.0020 0.0045496929 0.0001647394 0.06355928 0.014832948
C[C>A]C 0.0014 0.0037644739 0.0007368748 0.03375700 0.007822175
Signature.26 Signature.27 Signature.28 Signature.29 Signature.30
A[C>A]A 0.0020397729 0.0052056269 0.0013974388 0.06998199 0.0000000
A[C>A]C 0.0014871623 0.0047382274 0.0009171877 0.05515236 0.0000000
A[C>A]G 0.0002839456 0.0007826979 0.0000000000
0.01784698 0.0019673
A[C>A]T 0.0005978656 0.0027182425 0.0005134100 0.02680472 0.0000000
C[C>A]A 0.0037058501 0.0050650733 0.0011685156 0.05141021 0.0000000
C[C>A]C 0.0039807234 0.0022341533 0.0003342918 0.02582565 0.0000000
head(t(randomly.generated.tumors)[,1:5])
1 2 3 4 5
A[C>A]A 1.314202e-03 0.011030105 0.015149499 0.016360877 0.020639201
A[C>A]C 1.961651e-03 0.007874988 0.012403431 0.012688212 0.005849898
A[C>A]G 9.687434e-05 0.003877974 0.006080275 0.001582708 0.001652969
A[C>A]T 2.529105e-03 0.007840416 0.009771184 0.010520043 0.008789111
C[C>A]A 5.644115e-03 0.014087362 0.017718979 0.017555471 0.037798557
C[C>A]C 3.838037e-03 0.015529936 0.021613106 0.011758016 0.014110015
可以看到signatures.nature2013里面是对每个样本的96种突变情况算出了百分比,而signatures.cosmic是对30个signature的96种突变情况算出了百分比,随机生成的randomly.generated.tumors里面有100个样本。那么我们也应该对自己的数据算出这个96种突变情况算出了百分比。
算出96种突变情况算出了百分比
```bash
sigs.input <- mut.to.sigs.input(mut.ref = sample.mut.ref,
sample.id = "Sample",
chr = "chr",
pos = "pos",
ref = "ref",
alt = "alt")
class(sigs.input)
## [1] "data.frame"
head(t(sigs.input))
## 1 2
## A[C>A]A 9 1
## A[C>A]C 7 1
## A[C>A]G 5 0
## A[C>A]T 7 0
## C[C>A]A 10 3
## C[C>A]C 18 2
很明显,还是一个普通的数据框,这样的计算百分比其实难度很小,我在直播我的基因组上面也讲过。
推断signature的组成
# Determine the signatures contributing to the two example samples
sample_1 = whichSignatures(tumor.ref = sigs.input,
signatures.ref = signatures.nature2013,
sample.id = 1,
contexts.needed = TRUE,
tri.counts.method = 'default')
sample_2 = whichSignatures(tumor.ref = sigs.input,
signatures.ref = signatures.nature2013,
sample.id = 2,
contexts.needed = TRUE,
tri.counts.method = 'default')
这里的参考signature可以选择signatures.nature2013或者signatures.cosmic数据。
tri.counts.method可以选择exome,genome,exome2genome 来限定区域。
可视化
# Plot example
plot_example <- whichSignatures(tumor.ref = randomly.generated.tumors,
signatures.ref = signatures.nature2013,
sample.id = 13)
# Plot output
plotSignatures(plot_example, sub = 'example')
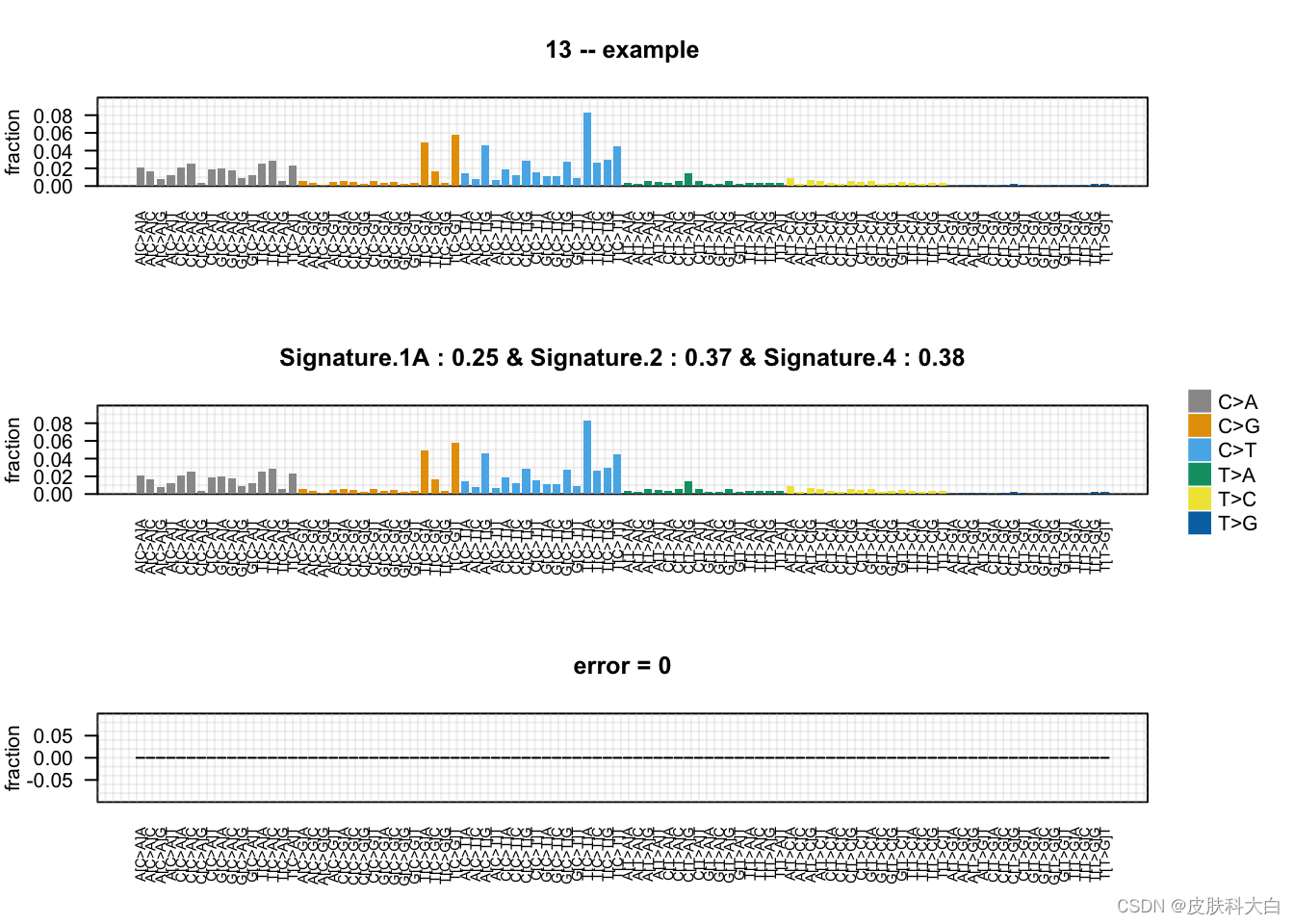
这里是挑出第13个样本,看看它的signature的组成。randomly.generated.tumors是随机生成的一个signature,里面有100个样本,不过要记住,这个数据集里面的样本,其实是一堆样本的意思。因为对单个样本算signature意义不大,单个样本,如果是WES,那么somatic mutation可能就百来个而已。