我们今天给大家演示下caret包做随机森林分类的一个小例子,同时也给大家看看做预处理和不做预处理两种情况下的模型表现。
加载R包和数据
rm(list = ls())
library(caret)
## Loading required package: ggplot2
## Loading required package: lattice
load(file = "../000机器学习/hotels_df.rdata")
str(hotels_df)
## tibble [75,166 × 10] (S3: tbl_df/tbl/data.frame)
## $ children : Factor w/ 2 levels "children","none": 2 2 2 2 2 2 2 2 2 2 ...
## $ hotel : Factor w/ 2 levels "City Hotel","Resort Hotel": 2 2 2 2 2 2 2 2 2 2 ...
## $ arrival_date_month : Factor w/ 12 levels "April","August",..: 6 6 6 6 6 6 6 6 6 6 ...
## $ meal : Factor w/ 5 levels "BB","FB","HB",..: 1 1 1 1 1 1 1 2 3 1 ...
## $ adr : num [1:75166] 0 0 75 75 98 98 107 103 145 97 ...
## $ adults : num [1:75166] 2 2 1 1 2 2 2 2 2 2 ...
## $ required_car_parking_spaces: Factor w/ 2 levels "none","parking": 1 1 1 1 1 1 1 1 1 1 ...
## $ total_of_special_requests : num [1:75166] 0 0 0 0 1 1 0 1 0 3 ...
## $ stays_in_week_nights : num [1:75166] 0 0 1 1 2 2 2 2 4 4 ...
## $ stays_in_weekend_nights : num [1:75166] 0 0 0 0 0 0 0 0 0 0 ...
这个数据一共有75166行,10列,数据维度不大,其中children这一列是结果变量,二分类,因子型,其余列都是预测变量。
结果变量两个分类间的差别还是很大的,可以看到大概是10:1的比例:
table(hotels_df$children)
##
## children none
## 6073 69093
咱们先做一个简单的探索性数据分析看看数据情况,就用咱们之前介绍过很多次的GGally包。
library(ggplot2)
library(GGally)
## Registered S3 method overwritten by 'GGally':
## method from
## +.gg ggplot2
ggbivariate(hotels_df, outcome = "children")+
scale_fill_brewer(type = "qual")

从这个图可以很清晰的看到结果变量的不平衡,预测变量中有很多是分类变量,几个数值型的预测变量好像在不同类别间的差别不是很大。
不做数据预处理
首先我们演示下不做数据预处理的情况,随机森林是一个“很包容”的算法,它对数据的要求非常低,不做预处理也是可以直接建立模型的。
下面我们直接开始,由于这个数据集不算小,所以运行很慢哈,内存小的电脑可能会直接卡死…
- 划分训练集、测试集,
- 重抽样方法选择10折交叉验证,
- 使用网格搜索,自定义网格范围,
- 在训练集建立模型。
一气呵成:
# 设定种子数
set.seed(3456)
# 根据结果变量的类别多少划分
trainIndex <- createDataPartition(hotels_df$children, p = 0.7,
list = FALSE)
head(trainIndex)
## Resample1
## [1,] 1
## [2,] 3
## [3,] 5
## [4,] 6
## [5,] 7
## [6,] 9
hotelsTrain <- hotels_df[ trainIndex,]
hotelsTest <- hotels_df[-trainIndex,]
dim(hotelsTrain)
## [1] 52618 10
dim(hotelsTest)
## [1] 22548 10
# 选择重抽样方法,10折交叉验证
trControl <- trainControl(method = "cv", number = 10,
classProbs = T,
summaryFunction = twoClassSummary
)
# 网格搜索,首先设定超参数范围
rfGrid <- expand.grid(mtry = seq(2,10,2),
splitrule = c( "gini", "extratrees", "hellinger"),
min.node.size = seq(1,15,2)
)
# 加速,CPU没这么多线程的改小一点
library(doParallel)
## Loading required package: foreach
## Loading required package: iterators
## Loading required package: parallel
cl <- makePSOCKcluster(16)
registerDoParallel(cl)
set.seed(8)
rffit1 <- train(x = hotelsTrain[,-1],
y = hotelsTrain$children,
method = "ranger",
trControl = trControl,
verbose = FALSE,
tuneGrid = rfGrid
)
rffit1
## Random Forest
##
## 52618 samples
## 9 predictor
## 2 classes: 'children', 'none'
##
## No pre-processing
## Resampling: Cross-Validated (10 fold)
## Summary of sample sizes: 47357, 47356, 47356, 47355, 47357, 47356, ...
## Resampling results across tuning parameters:
##
## mtry splitrule min.node.size ROC Sens Spec
## 2 gini 1 0.8768929 0.17827285 0.9952033
## 2 gini 3 0.8767803 0.17944932 0.9953066
## 2 gini 5 0.8761470 0.17685998 0.9955341
## 2 gini 7 0.8764686 0.17450925 0.9955341
## 2 gini 9 0.8759509 0.17451036 0.9956375
## 2 gini 11 0.8759252 0.17286219 0.9958235
## 2 gini 13 0.8759497 0.17004087 0.9958856
## 2 gini 15 0.8754730 0.16886772 0.9958235
## 2 extratrees 1 0.8675842 0.06773488 0.9992970
## 2 extratrees 3 0.8673029 0.06538470 0.9994624
## 2 extratrees 5 0.8672041 0.06373488 0.9995245
## 2 extratrees 7 0.8666000 0.06161944 0.9995451
## 2 extratrees 9 0.8664335 0.06020878 0.9995658
## 2 extratrees 11 0.8660962 0.05997349 0.9995038
## 2 extratrees 13 0.8659157 0.05550511 0.9995245
## 2 extratrees 15 0.8656270 0.05644573 0.9996278
## 2 hellinger 1 0.8785511 0.16275283 0.9962371
## 2 hellinger 3 0.8779519 0.16274896 0.9964025
## 2 hellinger 5 0.8779581 0.16228224 0.9962577
## 2 hellinger 7 0.8773762 0.15851754 0.9962784
## 2 hellinger 9 0.8774350 0.15992985 0.9963818
## 2 hellinger 11 0.8768638 0.15616791 0.9964231
## 2 hellinger 13 0.8769271 0.15334548 0.9965679
## 2 hellinger 15 0.8765844 0.15428445 0.9965059
## 4 gini 1 0.8766326 0.31373985 0.9860233
## 4 gini 3 0.8767241 0.30268600 0.9869950
## 4 gini 5 0.8773796 0.29610329 0.9880909
## 4 gini 7 0.8777288 0.28763325 0.9886491
## 4 gini 9 0.8781637 0.28457664 0.9894555
## 4 gini 11 0.8783674 0.27752168 0.9899103
## 4 gini 13 0.8787160 0.27187849 0.9900757
## 4 gini 15 0.8783649 0.26717371 0.9905099
## 4 extratrees 1 0.8758849 0.24365369 0.9902825
## 4 extratrees 3 0.8761048 0.23542171 0.9909648
## 4 extratrees 5 0.8762743 0.22789616 0.9924948
## 4 extratrees 7 0.8767513 0.21966529 0.9929910
## 4 extratrees 9 0.8767754 0.21425463 0.9934252
## 4 extratrees 11 0.8768805 0.20484618 0.9939627
## 4 extratrees 13 0.8769265 0.20061585 0.9945003
## 4 extratrees 15 0.8768345 0.19497045 0.9947898
## 4 hellinger 1 0.8785093 0.30715327 0.9867056
## 4 hellinger 3 0.8790078 0.30221431 0.9874706
## 4 hellinger 5 0.8793307 0.29868931 0.9881736
## 4 hellinger 7 0.8796394 0.29186854 0.9887732
## 4 hellinger 9 0.8797965 0.28457885 0.9893728
## 4 hellinger 11 0.8802855 0.28010936 0.9897449
## 4 hellinger 13 0.8803949 0.27493344 0.9899310
## 4 hellinger 15 0.8806475 0.27140679 0.9901585
## 6 gini 1 0.8725440 0.33960177 0.9822603
## 6 gini 3 0.8733587 0.33349020 0.9839558
## 6 gini 5 0.8741924 0.32196907 0.9854857
## 6 gini 7 0.8753984 0.31209279 0.9865402
## 6 gini 9 0.8756844 0.30574427 0.9874500
## 6 gini 11 0.8762144 0.30198012 0.9879669
## 6 gini 13 0.8758602 0.29703894 0.9882770
## 6 gini 15 0.8768813 0.28857332 0.9888972
## 6 extratrees 1 0.8712914 0.30057332 0.9835008
## 6 extratrees 3 0.8732889 0.28622535 0.9859819
## 6 extratrees 5 0.8742153 0.27493621 0.9876566
## 6 extratrees 7 0.8747571 0.26293952 0.9894762
## 6 extratrees 9 0.8755811 0.25259100 0.9904066
## 6 extratrees 11 0.8761412 0.24718255 0.9910682
## 6 extratrees 13 0.8767255 0.23706490 0.9916057
## 6 extratrees 15 0.8764662 0.23165866 0.9920192
## 6 hellinger 1 0.8748318 0.33466335 0.9832321
## 6 hellinger 3 0.8757301 0.33395857 0.9846794
## 6 hellinger 5 0.8766764 0.32549406 0.9859406
## 6 hellinger 7 0.8777220 0.31491466 0.9867883
## 6 hellinger 9 0.8780874 0.30973764 0.9874500
## 6 hellinger 11 0.8787693 0.30292129 0.9879462
## 6 hellinger 13 0.8788632 0.29704170 0.9884010
## 6 hellinger 15 0.8793159 0.29703949 0.9887525
## 8 gini 1 0.8697649 0.34736371 0.9802548
## 8 gini 3 0.8709826 0.34219111 0.9820950
## 8 gini 5 0.8714010 0.33184258 0.9839558
## 8 gini 7 0.8730885 0.32243634 0.9853204
## 8 gini 9 0.8732365 0.31350290 0.9862715
## 8 gini 11 0.8738872 0.30856393 0.9870985
## 8 gini 13 0.8744732 0.30456614 0.9874293
## 8 gini 15 0.8753895 0.29915714 0.9879048
## 8 extratrees 1 0.8710506 0.32220768 0.9800273
## 8 extratrees 3 0.8721929 0.30880309 0.9833561
## 8 extratrees 5 0.8737154 0.28974924 0.9864161
## 8 extratrees 7 0.8740291 0.27916708 0.9880288
## 8 extratrees 9 0.8751967 0.27399503 0.9890833
## 8 extratrees 11 0.8756311 0.26411709 0.9900344
## 8 extratrees 13 0.8758530 0.25588456 0.9906547
## 8 extratrees 15 0.8760888 0.24906435 0.9912129
## 8 hellinger 1 0.8725111 0.34078100 0.9824257
## 8 hellinger 3 0.8738181 0.33466667 0.9834802
## 8 hellinger 5 0.8751218 0.33207788 0.9849895
## 8 hellinger 7 0.8759385 0.32196631 0.9860647
## 8 hellinger 9 0.8763148 0.31232588 0.9867056
## 8 hellinger 11 0.8772747 0.30973875 0.9871812
## 8 hellinger 13 0.8774132 0.30621044 0.9877808
## 8 hellinger 15 0.8778141 0.30315438 0.9879669
## 10 gini 1 NaN NaN NaN
## 10 gini 3 NaN NaN NaN
## 10 gini 5 NaN NaN NaN
## 10 gini 7 NaN NaN NaN
## 10 gini 9 NaN NaN NaN
## 10 gini 11 NaN NaN NaN
## 10 gini 13 NaN NaN NaN
## 10 gini 15 NaN NaN NaN
## 10 extratrees 1 NaN NaN NaN
## 10 extratrees 3 NaN NaN NaN
## 10 extratrees 5 NaN NaN NaN
## 10 extratrees 7 NaN NaN NaN
## 10 extratrees 9 NaN NaN NaN
## 10 extratrees 11 NaN NaN NaN
## 10 extratrees 13 NaN NaN NaN
## 10 extratrees 15 NaN NaN NaN
## 10 hellinger 1 NaN NaN NaN
## 10 hellinger 3 NaN NaN NaN
## 10 hellinger 5 NaN NaN NaN
## 10 hellinger 7 NaN NaN NaN
## 10 hellinger 9 NaN NaN NaN
## 10 hellinger 11 NaN NaN NaN
## 10 hellinger 13 NaN NaN NaN
## 10 hellinger 15 NaN NaN NaN
##
## ROC was used to select the optimal model using the largest value.
## The final values used for the model were mtry = 4, splitrule = hellinger
## and min.node.size = 15.
我们之前已经铺垫了很多caret的基础知识,所以这里就不对结果做详细解读了,大家看不懂的去翻之前的推文吧。
最终选择的是mtry = 4, splitrule = hellinger and min.node.size = 15
下面随手再画个图:
ggplot(rffit1)+theme_bw()+theme(legend.position = "top")
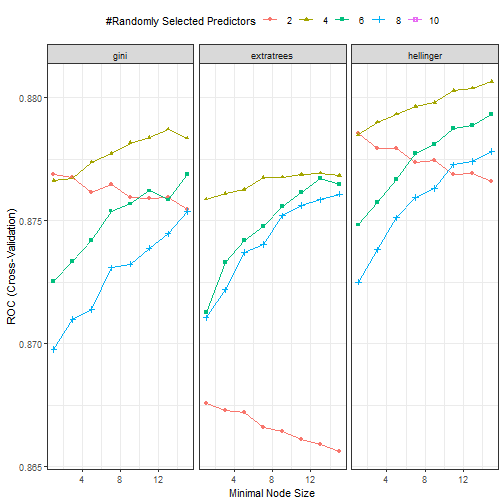
从这个图来看和上面的结果是一样的,mtry = 4, splitrule = hellinger and min.node.size = 15的时候,ROC是最高的,大概0.88多一点。
这个数据是不是很好了呢,还有进步的空间吗?不好说
下面我们对数据做一些常规的预处理,重新建立模型,再看一看效果。
做数据预处理
预处理
首先处理结果变量类不平衡的问题,我们这里就用downsampling吧,这个方法也在之前的推文中铺垫过了:R语言机器学习caret-06:重采样解决类不平衡
hotels <- downSample(x = hotels_df[,-1],
y = hotels_df$children,
yname = "Children"
)
table(hotels$Children)
##
## children none
## 6073 6073
Class <- hotels$Class
dim(hotels)
## [1] 12146 10
str(hotels)
## 'data.frame': 12146 obs. of 10 variables:
## $ hotel : Factor w/ 2 levels "City Hotel","Resort Hotel": 1 2 1 1 2 1 2 2 1 1 ...
## $ arrival_date_month : Factor w/ 12 levels "April","August",..: 6 6 4 3 8 2 6 4 7 1 ...
## $ meal : Factor w/ 5 levels "BB","FB","HB",..: 1 3 1 1 1 1 1 3 1 1 ...
## $ adr : num 185 186 198 0 77 ...
## $ adults : num 2 2 2 2 2 2 3 2 2 2 ...
## $ required_car_parking_spaces: Factor w/ 2 levels "none","parking": 2 1 1 1 1 2 1 1 1 1 ...
## $ total_of_special_requests : num 0 2 1 2 0 0 1 2 2 0 ...
## $ stays_in_week_nights : num 1 6 2 2 5 1 5 0 3 2 ...
## $ stays_in_weekend_nights : num 2 4 2 0 2 2 2 1 1 0 ...
## $ Class : Factor w/ 2 levels "children","none": 1 1 1 1 1 1 1 1 1 1 ...
这样处理后,结果变量的两个类基本一样多了,但是这个方法损失了很多信息哈,可以看到处理完只剩下12146行了…如果你的数据本身样本量就不大,就不要用这种方法了。
接下来对数值型变量去掉近零方差变量,并进行中心化和标准化,这几个操作可以一起进行:
zcs <- preProcess(hotels,
method = c("zv","center", "scale"))
hotels <- predict(zcs, newdata = hotels)
接下来我们对分类变量进行哑变量设置,这个哑变量的我们在之前也提到过很多次了,除了哑变量还有非常多的编码方式,大家感兴趣的去翻历史推文即可。
Children <- hotels$Children
dummy <- dummyVars(Children ~ ., data = hotels)
hotels <- predict(dummy, newdata = hotels)
hotels <- as.data.frame(hotels)
str(hotels)
## 'data.frame': 12146 obs. of 26 variables:
## $ hotel.City Hotel : num 1 0 1 1 0 1 0 0 1 1 ...
## $ hotel.Resort Hotel : num 0 1 0 0 1 0 1 1 0 0 ...
## $ arrival_date_month.April : num 0 0 0 0 0 0 0 0 0 1 ...
## $ arrival_date_month.August : num 0 0 0 0 0 1 0 0 0 0 ...
## $ arrival_date_month.December : num 0 0 0 1 0 0 0 0 0 0 ...
## $ arrival_date_month.February : num 0 0 1 0 0 0 0 1 0 0 ...
## $ arrival_date_month.January : num 0 0 0 0 0 0 0 0 0 0 ...
## $ arrival_date_month.July : num 1 1 0 0 0 0 1 0 0 0 ...
## $ arrival_date_month.June : num 0 0 0 0 0 0 0 0 1 0 ...
## $ arrival_date_month.March : num 0 0 0 0 1 0 0 0 0 0 ...
## $ arrival_date_month.May : num 0 0 0 0 0 0 0 0 0 0 ...
## $ arrival_date_month.November : num 0 0 0 0 0 0 0 0 0 0 ...
## $ arrival_date_month.October : num 0 0 0 0 0 0 0 0 0 0 ...
## $ arrival_date_month.September : num 0 0 0 0 0 0 0 0 0 0 ...
## $ meal.BB : num 1 0 1 1 1 1 1 0 1 1 ...
## $ meal.FB : num 0 0 0 0 0 0 0 0 0 0 ...
## $ meal.HB : num 0 1 0 0 0 0 0 1 0 0 ...
## $ meal.SC : num 0 0 0 0 0 0 0 0 0 0 ...
## $ meal.Undefined : num 0 0 0 0 0 0 0 0 0 0 ...
## $ adr : num 185 186 198 0 77 ...
## $ adults : num 2 2 2 2 2 2 3 2 2 2 ...
## $ required_car_parking_spaces.none : num 0 1 1 1 1 0 1 1 1 1 ...
## $ required_car_parking_spaces.parking: num 1 0 0 0 0 1 0 0 0 0 ...
## $ total_of_special_requests : num 0 2 1 2 0 0 1 2 2 0 ...
## $ stays_in_week_nights : num 1 6 2 2 5 1 5 0 3 2 ...
## $ stays_in_weekend_nights : num 2 4 2 0 2 2 2 1 1 0 ...
进行了这一步操作后预测变量明显变多了~
做完这一套操作后我们的数据变成了12146行和27列:
hotels$Children <- Children
str(hotels)
## 'data.frame': 12146 obs. of 27 variables:
## $ hotel.City Hotel : num 0.815 -1.227 0.815 0.815 -1.227 ...
## $ hotel.Resort Hotel : num -0.815 1.227 -0.815 -0.815 1.227 ...
## $ arrival_date_month.April : num -0.3 -0.3 -0.3 -0.3 -0.3 ...
## $ arrival_date_month.August : num -0.445 -0.445 -0.445 -0.445 -0.445 ...
## $ arrival_date_month.December : num -0.254 -0.254 -0.254 3.937 -0.254 ...
## $ arrival_date_month.February : num -0.277 -0.277 3.609 -0.277 -0.277 ...
## $ arrival_date_month.January : num -0.229 -0.229 -0.229 -0.229 -0.229 ...
## $ arrival_date_month.July : num 2.428 2.428 -0.412 -0.412 -0.412 ...
## $ arrival_date_month.June : num -0.294 -0.294 -0.294 -0.294 -0.294 ...
## $ arrival_date_month.March : num -0.283 -0.283 -0.283 -0.283 3.535 ...
## $ arrival_date_month.May : num -0.296 -0.296 -0.296 -0.296 -0.296 ...
## $ arrival_date_month.November : num -0.216 -0.216 -0.216 -0.216 -0.216 ...
## $ arrival_date_month.October : num -0.291 -0.291 -0.291 -0.291 -0.291 ...
## $ arrival_date_month.September : num -0.272 -0.272 -0.272 -0.272 -0.272 ...
## $ meal.BB : num 0.524 -1.907 0.524 0.524 0.524 ...
## $ meal.FB : num -0.0829 -0.0829 -0.0829 -0.0829 -0.0829 ...
## $ meal.HB : num -0.402 2.489 -0.402 -0.402 -0.402 ...
## $ meal.SC : num -0.254 -0.254 -0.254 -0.254 -0.254 ...
## $ meal.Undefined : num -0.096 -0.096 -0.096 -0.096 -0.096 ...
## $ adr : num 1.015 1.024 1.225 -1.959 -0.721 ...
## $ adults : num 0.236 0.236 0.236 0.236 0.236 ...
## $ required_car_parking_spaces.none : num -2.539 0.394 0.394 0.394 0.394 ...
## $ required_car_parking_spaces.parking: num 2.539 -0.394 -0.394 -0.394 -0.394 ...
## $ total_of_special_requests : num -0.976 1.189 0.106 1.189 -0.976 ...
## $ stays_in_week_nights : num -0.803 1.794 -0.284 -0.284 1.274 ...
## $ stays_in_weekend_nights : num 1.017 3.022 1.017 -0.988 1.017 ...
## $ Children : Factor w/ 2 levels "children","none": 1 1 1 1 1 1 1
建立模型
方法还是和之前一样的,就不多说了。
# 设定种子数
set.seed(3456)
# 根据结果变量的类别多少划分
trainIndex <- createDataPartition(hotels$Children, p = 0.7,
list = FALSE)
head(trainIndex)
## Resample1
## [1,] 1
## [2,] 2
## [3,] 3
## [4,] 5
## [5,] 6
## [6,] 7
hotelsTrain <- hotels[ trainIndex,]
hotelsTest <- hotels[-trainIndex,]
# 选择重抽样方法,10折交叉验证
trControl <- trainControl(method = "cv", number = 10,
classProbs = T,
summaryFunction = twoClassSummary
)
# 网格搜索,首先设定超参数范围
rfGrid <- expand.grid(mtry = seq(2,10,2),
splitrule = c( "gini", "extratrees", "hellinger"),
min.node.size = seq(1,15,2)
)
# 设置种子数,进行建模
library(doParallel)
cl <- makePSOCKcluster(16)
registerDoParallel(cl)
set.seed(8)
rffit <- train(x = hotelsTrain[,-15],
y = hotelsTrain$Children,
method = "ranger",
trControl = trControl,
verbose = FALSE,
#tuneLength = 10
tuneGrid = rfGrid
)
rffit
## Random Forest
##
## 8504 samples
## 26 predictor
## 2 classes: 'children', 'none'
##
## No pre-processing
## Resampling: Cross-Validated (10 fold)
## Summary of sample sizes: 7654, 7654, 7654, 7652, 7654, 7653, ...
## Resampling results across tuning parameters:
##
## mtry splitrule min.node.size ROC Sens Spec
## 2 gini 1 1 1 1
## 2 gini 3 1 1 1
## 2 gini 5 1 1 1
## 2 gini 7 1 1 1
## 2 gini 9 1 1 1
## 2 gini 11 1 1 1
## 2 gini 13 1 1 1
## 2 gini 15 1 1 1
## 2 extratrees 1 1 1 1
## 2 extratrees 3 1 1 1
## 2 extratrees 5 1 1 1
## 2 extratrees 7 1 1 1
## 2 extratrees 9 1 1 1
## 2 extratrees 11 1 1 1
## 2 extratrees 13 1 1 1
## 2 extratrees 15 1 1 1
## 2 hellinger 1 1 1 1
## 2 hellinger 3 1 1 1
## 2 hellinger 5 1 1 1
## 2 hellinger 7 1 1 1
## 2 hellinger 9 1 1 1
## 2 hellinger 11 1 1 1
## 2 hellinger 13 1 1 1
## 2 hellinger 15 1 1 1
## 4 gini 1 1 1 1
## 4 gini 3 1 1 1
## 4 gini 5 1 1 1
## 4 gini 7 1 1 1
## 4 gini 9 1 1 1
## 4 gini 11 1 1 1
## 4 gini 13 1 1 1
## 4 gini 15 1 1 1
## 4 extratrees 1 1 1 1
## 4 extratrees 3 1 1 1
## 4 extratrees 5 1 1 1
## 4 extratrees 7 1 1 1
## 4 extratrees 9 1 1 1
## 4 extratrees 11 1 1 1
## 4 extratrees 13 1 1 1
## 4 extratrees 15 1 1 1
## 4 hellinger 1 1 1 1
## 4 hellinger 3 1 1 1
## 4 hellinger 5 1 1 1
## 4 hellinger 7 1 1 1
## 4 hellinger 9 1 1 1
## 4 hellinger 11 1 1 1
## 4 hellinger 13 1 1 1
## 4 hellinger 15 1 1 1
## 6 gini 1 1 1 1
## 6 gini 3 1 1 1
## 6 gini 5 1 1 1
## 6 gini 7 1 1 1
## 6 gini 9 1 1 1
## 6 gini 11 1 1 1
## 6 gini 13 1 1 1
## 6 gini 15 1 1 1
## 6 extratrees 1 1 1 1
## 6 extratrees 3 1 1 1
## 6 extratrees 5 1 1 1
## 6 extratrees 7 1 1 1
## 6 extratrees 9 1 1 1
## 6 extratrees 11 1 1 1
## 6 extratrees 13 1 1 1
## 6 extratrees 15 1 1 1
## 6 hellinger 1 1 1 1
## 6 hellinger 3 1 1 1
## 6 hellinger 5 1 1 1
## 6 hellinger 7 1 1 1
## 6 hellinger 9 1 1 1
## 6 hellinger 11 1 1 1
## 6 hellinger 13 1 1 1
## 6 hellinger 15 1 1 1
## 8 gini 1 1 1 1
## 8 gini 3 1 1 1
## 8 gini 5 1 1 1
## 8 gini 7 1 1 1
## 8 gini 9 1 1 1
## 8 gini 11 1 1 1
## 8 gini 13 1 1 1
## 8 gini 15 1 1 1
## 8 extratrees 1 1 1 1
## 8 extratrees 3 1 1 1
## 8 extratrees 5 1 1 1
## 8 extratrees 7 1 1 1
## 8 extratrees 9 1 1 1
## 8 extratrees 11 1 1 1
## 8 extratrees 13 1 1 1
## 8 extratrees 15 1 1 1
## 8 hellinger 1 1 1 1
## 8 hellinger 3 1 1 1
## 8 hellinger 5 1 1 1
## 8 hellinger 7 1 1 1
## 8 hellinger 9 1 1 1
## 8 hellinger 11 1 1 1
## 8 hellinger 13 1 1 1
## 8 hellinger 15 1 1 1
## 10 gini 1 1 1 1
## 10 gini 3 1 1 1
## 10 gini 5 1 1 1
## 10 gini 7 1 1 1
## 10 gini 9 1 1 1
## 10 gini 11 1 1 1
## 10 gini 13 1 1 1
## 10 gini 15 1 1 1
## 10 extratrees 1 1 1 1
## 10 extratrees 3 1 1 1
## 10 extratrees 5 1 1 1
## 10 extratrees 7 1 1 1
## 10 extratrees 9 1 1 1
## 10 extratrees 11 1 1 1
## 10 extratrees 13 1 1 1
## 10 extratrees 15 1 1 1
## 10 hellinger 1 1 1 1
## 10 hellinger 3 1 1 1
## 10 hellinger 5 1 1 1
## 10 hellinger 7 1 1 1
## 10 hellinger 9 1 1 1
## 10 hellinger 11 1 1 1
## 10 hellinger 13 1 1 1
## 10 hellinger 15 1 1 1
##
## ROC was used to select the optimal model using the largest value.
## The final values used for the model were mtry = 2, splitrule = gini
## and min.node.size = 1.
结果很明显比不做预处理好多了,ROC直接到1了!而且由于我们用了downsampling,运行速度明显加快。
下面还是画个图更直观的看一下:
ggplot(rffit)+theme_bw()+theme(legend.position = "top")

效果还是很显著的,这样我们就选择一个最简单的模型就可以了。看看caret包为我们自动选择的最终模型:
rffit$finalModel
## Ranger result
##
## Call:
## ranger::ranger(dependent.variable.name = ".outcome", data = x, mtry = min(param$mtry, ncol(x)), min.node.size = param$min.node.size, splitrule = as.character(param$splitrule), write.forest = TRUE, probability = classProbs, ...)
##
## Type: Probability estimation
## Number of trees: 500
## Sample size: 8504
## Number of independent variables: 26
## Mtry: 2
## Target node size: 1
## Variable importance mode: none
## Splitrule: gini
## OOB prediction error (Brier s.): 0.04232395
查看自动为我们选择的超参数:
rffit$bestTune
## mtry splitrule min.node.size
## 1 2 gini 1
训练集
然后就可以直接用这个模型在测试集做预测了,我这里直接把测试集的真实类别,预测类别,预测概率放在一个数据框中了,方便进行各种计算和操作。
library(dplyr)
##
## Attaching package: 'dplyr'
## The following objects are masked from 'package:stats':
##
## filter, lag
## The following objects are masked from 'package:base':
##
## intersect, setdiff, setequal, union
test_pred <- select(hotelsTest, Children) %>%
bind_cols(predict(rffit, newdata = hotelsTest,type = "prob")) %>%
bind_cols(pred = predict(rffit, newdata = hotelsTest))
head(test_pred)
## Children children none pred
## 4 children 0.7915756 0.2084244 children
## 12 children 0.8770473 0.1229527 children
## 14 children 0.8565740 0.1434260 children
## 19 children 0.7310673 0.2689327 children
## 23 children 0.8696240 0.1303760 children
## 25 children 0.8097900 0.1902100 children
混淆矩阵:
confusionMatrix(data = test_pred$Children, test_pred$pred)
## Confusion Matrix and Statistics
##
## Reference
## Prediction children none
## children 1821 0
## none 0 1821
##
## Accuracy : 1
## 95% CI : (0.999, 1)
## No Information Rate : 0.5
## P-Value [Acc > NIR] : < 2.2e-16
##
## Kappa : 1
##
## Mcnemar's Test P-Value : NA
##
## Sensitivity : 1.0
## Specificity : 1.0
## Pos Pred Value : 1.0
## Neg Pred Value : 1.0
## Prevalence : 0.5
## Detection Rate : 0.5
## Detection Prevalence : 0.5
## Balanced Accuracy : 1.0
##
## 'Positive' Class : children
##
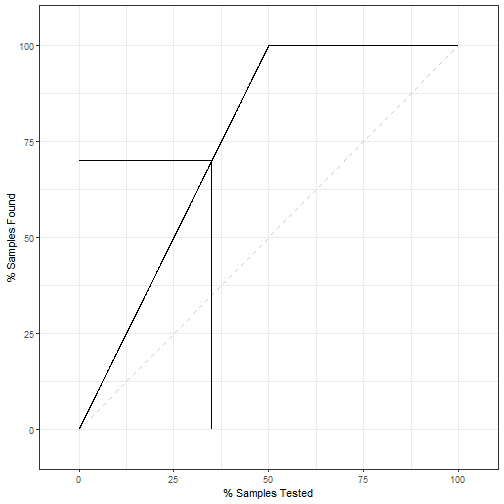
提升曲线:
lift_obj <- lift(Children ~ children, data = test_pred)
ggplot(lift_obj,values = 70)+theme_bw()
校准曲线:
cal_obj <- calibration(Children ~ children, data = test_pred,cuts = 10)
ggplot(cal_obj)+theme_bw()
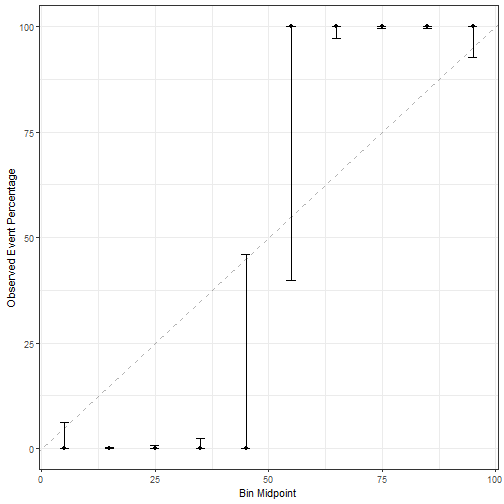
可以看出我们的模型区分度很好,但是校准度一塌糊涂。
多个模型的比较我们之前也演示过了,大家可以参考之前的推文。